作者简介:王丽(1981-),女,甘肃兰州人,讲师,在读博士。E-mail:39726821@qq.com
以NaCl胁迫处理的拟南芥幼苗叶片为材料,用RNA提取试剂盒抽提总RNA,通过RT-PCR技术和DNA序列测定分析,证实获得了拟南芥高亲和性K+载体蛋白基因( AtHKT1)的cDNA序列。该cDNA全长1521 bp,包括506个氨基酸和1个终止密码子序列,且与原序列(accession number AF237672)同源性为99.34%,但与其他科植物 HKT1基因同源性较低,注册该基因到GenBank中,注册号为AY685182。利用生物信息学相关软件分析预测 AtHKT1基因蛋白质功能和结构,结果发现,该蛋白分子量为57.45 kD,理论等电点为9.33;氨基酸序列中第1~40个氨基酸属信号肽序列;第152~500个氨基酸属Trk H阳离子转运体蛋白保守结构域,并存在蛋白激酶C,酪氨酸蛋白激酶,依赖cAMP/cGMP蛋白激酶磷酸化,糖基化和豆蔻酰化等功能位点;该基因编码的蛋白有10个跨膜结构,N末端、C末端及中部等多个跨膜区具疏水性,符合载体类运输蛋白特点。表明本研究获得了拟南芥 AtHKT1基因。
The full length coding sequence of AtHKT1 was cloned via RT-PCR (reverse transcription-polymerase chain reaction) from the leaves of Arabidopsis thaliana seedling under NaCl stress. The full-length cDNA contained 1521 bp and encoded a polypeptide of 506 amino acids and a termination codon. The sequence shared 99.34% homology with the reported sequence (accession number AF237672), but the homologies were very low compared with similar genes from other plants. The gene was submitted to GenBank and was given the registration number AY685182. Bioinformatics softwares were used to predict and analyze the protein function and structure of the AtHKT1 gene cDNA sequence. The molecular weight of the deduced polypeptide of AtHKT1 was 57.45 kD. The theoretical pI was 9.33. The N-terminal amino acids from 1 to 40 formed a classic signal peptide sequence. The peptide from 152 to 500 was a Trk H cation transporter conserved domain. It might also carry protein kinase C, tyrosine kinase, cAMP-and cGMP-dependent protein kinase phosphorylation, glycosylation, N-myristoylation sites and other function sites. The deduced peptide contained 10 transmembrane domains. The transmembrane domains near the N-terminus, the C-terminus and the middle part of the peptide displayed high hydrophobicity. Therefore, it is suggested that the gene product of AtHKT1 might play roles in facilitating cation transportation.
钾是植物生长发育必需的大量元素, 参与植物许多生理生化过程, 包括酶活性调节、蛋白质合成及渗透调节等, 保持胞质内较高K+/Na+对植物生长起着非常重要的作用[1]。盐胁迫时, 大量Na+从根部进入植物体内, 抑制K+的吸收, K+/Na+降低, 导致植物受害[2]。研究表明, 植物的耐盐性与其维持体内K+、Na+平衡的能力密切相关, 具有高K+/Na+的植物有较强的耐盐性[3]。因此, 限制Na+吸收、增加Na+外排, 同时保证K+吸收, 维持细胞质高K+/Na+可抵御过量的Na+对植物造成的伤害[4]。HKT(high-affinity K+ transporter)类蛋白属于一个广泛存在于植物、真菌、真细菌和古细菌中的K+转运体超级家族, 具有可以调节植物体内K+/Na+平衡的能力[5]。
自1994年Schachtman和Schroeder[6]从小麦(Triticum aestivum)幼苗根cDNA表达文库中克隆到第1个高亲和性K+载体蛋白基因-TaHKT1以来, 陆续在拟南芥(Arabidopsis thaliana)[7]、碱蓬(Suaeda salsa)[5]、芦苇(Phragmites australis)[8]、水稻(Oryza sativa)[9]、冰叶日中花(Mesembryanthemum crystallinum)[10]、桉树(Eucalyptus camaldulensis)[11]、大麦(Hordeum vulgare)[12]、商陆(Phytolacca acinosa)[13]、海蓬子(Salicornia bigelovii)[14]等多种植物中克隆了其同源蛋白基因。早期研究认为, HKT1蛋白是Na+吸收和运输的载体, 降低HKT1的表达, 可能会提高植物抗盐性, 如Laurie等[15]研究发现, 表达反义HKT1基因的小麦植株较野生型耐盐胁迫能力更强; Horie等[16]比较耐盐水稻和盐敏感水稻时发现, 抗盐性高的水稻HKT1基因的表达量在盐胁迫时降低, 而盐敏感型水稻HKT1的表达量一直维持在很高水平; Rus等[17]研究发现拟南芥AtHKT1基因在sos3-1突变体的过量表达增强了突变体植株在低K+环境下对Na+的敏感性及植株的缺K+症状, AtHKT1基因可转运Na+进入细胞、调控根部Na+的吸收。然而近期更多的研究证明, AtHKT1转运蛋白在提高植物耐盐性中可能具有重要的积极作用, 如Mä ser等[18]认为拟南芥AtHKT1基因参与调控Na+在根和地上部分的分配; Berthomieu等[19]认为AtHKT1参与Na+通过韧皮部汁液从地上部分至根部的再循环过程, 降低叶中Na+积累; Sunarpi等[20]认为在高盐环境下, AtHKT1基因调节Na+从根部木质部卸载。特别是最近的研究又发现, AtHKT1基因既不调控Na+从地上部到根部的再循环, 也不调节Na+以内流的形式进入根部, 而是将Na+直接滞留在根的木质部中, 加载Na+进入根部液泡, 从而降低Na+从根到茎的运输[21, 22, 23, 24, 25, 26]。由此可见, AtHKT1基因在植株中的耐盐表达调控机制仍有争议, 目前尚未见利用该基因改良作物耐盐性的报道。
为了研究AtHKT1基因参与植物耐盐调控机制, 更好地发挥其作用, 本研究以拟南芥为材料, 利用RT-PCR技术, 在盐胁迫条件下进行AtHKT1基因的分离克隆。进一步通过该基因编码蛋白特性及结构分析, 初步确定该基因功能和特点, 为进一步研究该基因功能及改良作物耐盐性奠定基础。
1.1.1 植物材料 拟南芥种子用70%酒精浸泡30 s, 0.1% HgCl2处理5 min, 无菌水冲洗5次后接入MS基本培养基上, 在25℃、12 h/d光照下培养。当幼苗生长至14 d时, 将160 mmol/L的NaCl水溶液倒入培养基上, 胁迫幼苗12 h待用。本试验在2004年进行。
1.1.2 菌株和载体 大肠杆菌(Escherichia coli)菌株为DH5α 的感受态细胞; T-载体为pGEM-T Easy Vector(购自Promega公司), 抗性标记为氨苄青霉素(Ampr)。
1.1.3 酶和试剂 SuperscriptⅡ 逆转录试剂盒购自Promega公司; 各种限制性内切酶、PyrobestTMDNA聚合酶和T4DNA连接酶购自TaKaRa公司; TaqDNA聚合酶和TRIzol一步法RNA提取试剂盒购自鼎国公司; DNA凝胶回收试剂盒购自上海开瑞公司; 其他生化试剂为国产分析纯。
1.2.1 逆转录合成cDNA第1链 1)总RNA提取。用TRIzol一步法RNA提取试剂盒抽提盐胁迫下的拟南芥叶片总RNA, 提取方法见试剂盒说明书。提取的总RNA在经焦碳酸二乙酯(DEPC)处理的1%琼脂糖凝胶上电泳, EB染色后检查RNA完整性; 2)cDNA第1链合成。在提取的7.5 μ L RNA中加入1 μ L Oligo(dT), 65℃加热8 min, 室温自然冷却; 向上述混合液中加入4 μ L 25 mmol/L的MgCl2、4 μ L 5× buffer、2 μ L 10 mmol/L dNTP和0.5 μ L的RNase, 混匀后加入AMV反转录酶, 再次混匀后42℃下保温30 min, 99℃下煮5 min, -20℃保存。
1.2.2 PCR扩增 根据已发表的拟南芥AtHKT1基因序列(accession number AF237672), 设计1对引物, 即上游引物p1:5'-CGGGATCC(BamHⅠ )ATGGACAGAGTGGTGGCAAAAATAG-3'和下游引物p2:5'-CGGAGCTC(SacⅠ )TTAGGAAGACGAGGGGTAAAGTATCC-3', 由北京赛百盛公司合成。扩增反应体系为25 μ L, 由1 μ L cDNA模板、2.5 μ L 10× buffer、1 μ L dNTP(10 mmol/L)、1 μ L引物p1(10 μ mol/L)、1 μ L引物p2(10 μ mol/L)、0.5 μ L PyrobestTMDNA聚合酶、18 μ L dd H2O组成。扩增条件为:94℃预变性1 min后, 进行94℃ 50 s、53℃ 50 s、72℃ 120 s的35个循环, 再在72℃下延伸10 min。扩增产物EB染色后进行1%琼脂糖凝胶电泳检测。
1.2.3 AtHKT1基因cDNA与T-载体连接 扩增产物经1%琼脂糖凝胶电泳分离后回收, 回收方法见上海开瑞公司凝胶回收试剂盒说明书。扩增产物利用TaqDNA聚合酶对其加A, 然后与T-载体相连, 连接产物转化感受态细胞。
将转化细胞涂在含50 μ g/mL Ampr、20 mg/mL 5-溴-4-氯-3-吲哚-β -D-半乳糖苷(X-gal)和100 mg/mL异丙基-β -D-硫代半乳糖苷(IPTG)的LB平板上进行蓝白斑筛选。对所获得的白斑通过滞后质粒、PCR扩增、BamHⅠ 和SacⅠ 双酶切鉴定, -80℃保存菌株。
送鉴定后的白斑菌株到上海博亚公司测序, 所测序列用DNAMAN virsion 6.0软件进行分析。然后通过NCBI网站的Blast X和Blast P程序进行序列相似性分析; 利用 InterProScan (http://www.ebi.ac.uk/Tools/InterProScan/)进行信号肽分析和氨基酸保守性预测; 用MEGA4.0软件进行氨基酸序列比对和HKT类蛋白质序列系统进化树分析; 链接至http://expasy.org/tools/protparam.html网站进行蛋白质理化性质分析; 利用PredictProtein Server(http://www.predictprotein.org/)进行蛋白质结构域分析; 利用TMHMM Serverv 2.0(http://www.cbs.dtu.dk/services/TMHMM-2.0)程序分析蛋白质序列跨膜区; 利用ProtScale(http://expasy.org/tools/protscale.html)分析氨基酸序列的疏水性。
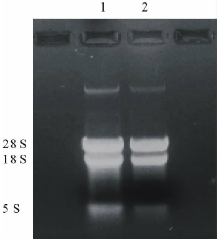
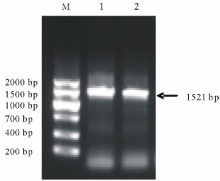
利用TRIzol一步法RNA提取试剂盒提取盐胁迫下的拟南芥叶片总RNA, 琼脂糖凝胶电泳显示, 总RNA 5S、18S和28S三条带清晰、完整(图1), 说明RNA完整性良好。利用RT-PCR试剂盒进行cDNA第1链合成后, 利用1对特异性引物及PyrobestTMDNA聚合酶, 以cDNA为模板进行目的基因扩增, 期望获得约1521 bp大小的DNA片段。对扩增产物电泳检查结果表明, 扩增产物与预期片段的大小基本一致(图2), 说明可能获得了AtHKT1基因的cDNA克隆。
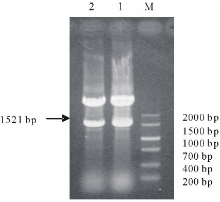
因pGEM-T Easy Vector载体多克隆位点位于LacZ基因编码区, 外源基因插入将改变LacZ基因编码序列的完整性, 影响β -半乳糖苷酶活性, 致使重组子在X-gal/IPTG平板上呈白色, 而非重组子呈蓝色。选取白色菌株进行滞后质粒筛选(图3), 初步淘汰假阳性, 然后对阳性菌株进行PCR(图4)及双酶切(BamHⅠ 和SacⅠ )鉴定(图5)。结果显示, 扩增到或切下约1521 bp大小的DNA片段, 说明可能获得了携带有AtHKT1基因的重组菌株。
 | 图4 白斑滞后质粒PCR鉴定Fig.4 Identification of white color plasmid by PCRM:MakerⅤ 分子量标记DNA marker Ⅴ ; 1~5:白斑滞后质粒White color plasmid. |
2.3.1 cDNA序列特征分析 将经鉴定的重组菌株交上海博亚生物技术公司测序。将测序结果与原序列(accession number AF237672)比较, 发现二者同源性高达99.34%。对测序结果分析后发现, 其开放阅读框架为1521 bp(图6), 包括506个氨基酸编码区和1个终止密码子(TAA)。该基因登记在GenBank中(accession number AY685182)。
用DNAMAN软件对该序列与GenBank中登记的部分物种的HKT1基因的cDNA序列进行同源性比较。结果发现, 该序列与其他学者登录的拟南芥AtHKT1基因同源性都很高, 达99.34%以上, 与同科盐芥同源性较高, 为84.00%, 与其他科植物同源性低, 为48.47%~57.34%(表1), 说明同科植物特别是同种植物, 该基因的保守性强, 而不同科植物的AtHKT1基因间差异较大。
2.3.2 蛋白质进化特征分析 系统进化树分析发现, 10个物种的21个HKT蛋白被聚为9个类群, 拟南芥AtHKT1和同科的盐芥ThHKT1聚为一类, 亲缘关系最近; 禾本科水稻OsHKT3、OsHKT9、OsHKT7、OsHKT4聚为一类; 禾本科水稻OsHKT1、OsHKT2和芦苇PhaHKT1为一类; 禾本科大麦HvHKT1和小麦TaHKT1为一类; 番杏科冰花McHKT2、桃金娘科赤桉EcHKT1、EcHKT2和藜科碱蓬SsHKT1为一类; 禾本科水稻OsHKT8、OsHKT6、番杏科冰花McHKT1、藜科海蓬子SbHKT1单独聚为一类, 基本表现为同科尤其是同种同源性高、种间相对低的特点(图7)。
2.3.3 蛋白理化性质及结构域分析 Protparam软件预测AtHKT1 蛋白分子式为C2657H4126N648O725S22, 分子量57.45 kD, 理论等电点为9.33, 带正电荷氨基酸残基数(Arg+Lys)为47, 带负电荷氨基酸残基数(Asp+Glu)为41, 不稳定系数为34.19, 为稳定碱性蛋白。该蛋白在第1~40个氨基酸残基处有1个信号肽序列(图6, 用“ · ” 表示)。在第152~500个氨基酸处有1个Trk H阳离子转运体蛋白保守结构域(图6, 用“ [ ]” 表示)。分别在第70~73, 128~131, 138~141, 185~188, 191~194, 260~263, 318~321, 339~342, 411~414个氨基酸残基处有9个酪蛋白激酶Ⅱ 磷酸化位点(图6, 用“ □” 表示), 表明该蛋白是一种真核细胞中普遍存在的信使非依赖性丝/苏氨酸蛋白激酶, 在细胞生存、生长、增殖及凋亡过程中具有重要功能。在第48~50, 185~187, 191~193, 260~262, 282~284, 413~415, 492~494和495~497个氨基酸处具有8个蛋白激酶C磷酸化位点(图6, 用“ ■” 表示), 能将靶蛋白的Ser/thr磷酸化, 从而激活细胞质中某些酶的活性, 参与生化反应调控; 还可与核中转录因子相互作用, 参与基因表达调控。该蛋白在第225~228个氨基酸处有1个依赖cAMP/cGMP蛋白激酶磷酸化位点[图6, 用“ ( )” 表示], 能在ATP存在的情况下使许多蛋白质特定的丝/苏氨酸残基磷酸化, 调节细胞的物质代谢和基因表达。此外, 在第261~267个氨基酸处[图6, 用“ { }” 表示], 该蛋白具有1个酪氨酸蛋白激酶磷酸化位点, 可将自身的酪氨酸磷酸化或将各种不同靶蛋白的酪氨酸磷酸化来实现信号传递, 其转导的信号通常与细胞生长、繁殖、分化、生存有关。位于第429~432个氨基酸处该蛋白具有1个N端糖基化位点(图6, 用“ =” 表示), 使蛋白质能够抵抗消化酶的作用、赋予蛋白质传导信号的功能、或某些蛋白只有在糖基化之后才能正确折叠。此外, 3个N端豆蔻酰化位点分别在第122~127, 394~399, 459~464个氨基酸处(图6, 用“ -” 表示)。
 | 图6 拟南芥AtHKT1基因的cDNA序列及其推测的氨基酸序列Fig.6 Nucleotide sequence and deduced amino sequence of AtHKT1 cDNA of A. thaliana |
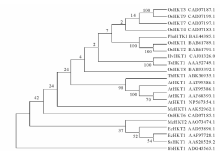 | 图7 AtHKT1 蛋白与其他物种 HKT 类蛋白构建的系统进化树分析Fig.7 Phylogenetic tree analysis of AtHKT1 form A. thaliana with other reported HKT proteins |
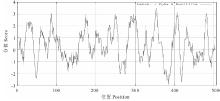
2.3.4 蛋白跨膜性及疏水性分析 经TMHMM Serverv 2.0软件分析推测, AtHKT1 蛋白有10个跨膜区(21~43, 82~104, 158~180, 195~217, 230~252, 285~307, 343~365, 395~412, 425~447, 467~486), 其中395~412区域的跨膜螺旋长度为18个氨基酸残基, 467~486区域的跨膜螺旋长度为20个氨基酸残基, 其余区域均为23个氨基酸残基(图8)。利用ProtScale软件的Kyte and Doolittle算法对AtHKT1蛋白进行亲水/疏水性分析(图9)。在347~359区域的疏水性最强(最大值达3.467), 疏水性较强的区域有13个, 分别位于第22~39, 58~68, 80~93, 157~164, 170~181, 199~206, 231~238, 245~256, 283~306, 326~328, 397~411, 430~434, 476~484个氨基酸处; 在372~390区域的亲水性最强(分值为-2.900), 其次有5个区域(45~52, 127~131, 139~141, 259~276, 415~422)具有较强的亲水性。就整体来看, 该蛋白疏水性氨基酸均匀分布在整个肽链中, 占总氨基酸组成的46.64%, 疏水区域大于亲水区域, N末端、C末端及中部等多个跨膜区均具有疏水性, 整个多肽链表现为疏水性, 可认为拟南芥AtHKT1蛋白是疏水性蛋白, 符合载体类运输蛋白特点, 表明获得了跨膜运输蛋白基因— — AtHKT1基因。
| 表1 拟南芥AtHKT1基因cDNA与其他植物HKT1基因cDNA序列同源性比较 Table 1 Alignment of nucleotide sequence of AtHKT1 gene with other homologues |
已有研究表明AtHKT1基因参与Na+的转运, 与植物耐盐性有关, 但关于其具体功能, 不同研究者存在着不同观点。早期研究认为, AtHKT1基因的过量表达可增加植株的盐敏感性[17], 近期部分学者对该基因的作用有新解, 认为AtHKT1基因可调节Na+在根和茎中的分配以及从茎到根部的再循环[18, 19, 20], AtHKT1蛋白如果在拟南芥根部的中柱鞘及维管束过量表达, 会增加Na+在根部木质部薄壁组织及皮层中滞留, 减少Na+由根部转运至叶, 从而降低叶片中Na+浓度, 表明该基因在根部的过量表达可提高拟南芥植株的耐盐性, 但如果由组成型35S启动子驱动AtHKT1基因在整个植株中的过量表达却导致了转基因植株的盐敏感性, 说明AtHKT1基因的组织特异性表达与植株耐盐性关系密切[24, 25, 26]。本研究根据GenBank中拟南芥高亲和性K+载体蛋白基因序列(AF237672)设计引物, 利用RT-PCR方法, 从拟南芥总RNA中扩增出目的cDNA片段。序列分析发现, 该cDNA片段的开放阅读框架为1521 bp, 包括506个氨基酸编码区和1个终止密码子(TAA)。该片段与GenBank中其他学者发表的拟南芥该基因cDNA序列(NM_117099)同源性为99.67%, 与同科盐芥(EF025500)cDNA同源性为84.00%, 蛋白质同源性为78%, 与其他科植物cDNA序列同源性较低, 为48.47%~57.34%, 蛋白质序列同源性为42.00%~51.00%。说明同一物种中该基因保守, 但不同物种间差异明显, 这与其他研究者对不同基因或不同物种同源性分析时的结论是一致的, 即属内同源性高, 属间相对低[27, 28, 29], 可见高亲和性K+载体蛋白在进化上也存在多样性。
本研究对克隆到的AtHKT1基因的cDNA片段利用相关软件进行了分析, 发现其具有1个Trk H阳离子转运体蛋白保守结构域和1个信号肽序列, 存在蛋白激酶C、酪氨酸蛋白激酶、酪蛋白激酶Ⅱ 磷酸化、依赖cAMP/cGMP蛋白激酶磷酸化、糖基化、豆蔻酰化等功能位点, 表明该基因具有阳离子转运功能。王景艳等[30]认为植物通过离子外排和区域化降低Na+的毒害与植物跨膜运输蛋白有密切关系, AtHKT1蛋白疏水性及跨膜性分析表明, 该蛋白有10个跨膜区, 是疏水性蛋白, 符合载体类跨膜运输蛋白特点, 推测其与钾和钠离子的转运相关。本研究为该基因进一步功能研究奠定了基础。
The authors have declared that no competing interests exist.
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|
| [30] |
|