作者简介:顾晓燕(1989-),女,甘肃兰州人,在读硕士。E-mail:mobei_ttxs@126.com
利用SRAP标记,对来自亚洲的84份老芒麦种质的遗传多样性和遗传关系进行了分析。23个引物组合共产生337条扩增带,其中203条为多态性带,多态性比率为60.24%。各种质间遗传相似系数的变幅为0.783~0.965,平均值为0.865。来自于青藏高原和蒙古的种质间的平均遗传相似性(GS)值最小(0.830),而来自于俄罗斯和蒙古的种质间的平均GS值最大(0.897)。对84份种质的聚类分析表明,供试种质可以划分成2大类,而且聚类结果与原始相似性矩阵间具有很高的吻合度(
SRAP markers were used to analyze genetic diversity and genetic relationship among eighty-four
研究和了解植物种质间的遗传变异,对于其在育种上的有效利用非常有益。根据遗传变异的信息,可以确定具有优异农艺性状的亲本组合[ 1],拓宽植物品种的遗传基础,防止其育种进程中的渐进性遗传衰退。另外,遗传多样性还可以为物种保护计划的制定和实施提供参考和借鉴。
老芒麦( Elymus sibiricus),别名西伯利亚野麦草,是披碱草属的模式种,为多年生、疏丛型、自花授粉的异源四倍体禾草,具有StStHH的染色体组构成[ 2]。它是欧亚大陆的广布种,在北美的阿拉斯加和加拿大北部也有少量分布[ 3]。老芒麦一般生长于湿润的草地、河滩、灌木丛中或森林边缘地带。在中亚及中国新疆、内蒙古和青藏高原地区,老芒麦作为一种重要的牧草在草地畜牧业中发挥了巨大作用[ 4, 5, 6]。
目前对老芒麦种质的遗传多样性研究很少。袁庆华等[ 7]发现21份老芒麦种质在13个农艺和形态学性状上存在高度变异。但形态性状具有不少缺点,如数目有限、易受环境影响和非均匀分布等。DNA分子标记的发展,极大地增加了研究遗传多样性的方法,它可以揭示整个基因组上可能存在的大量变异[ 8]。序列相关扩增多态性(sequence related amplified polymorphisms,SRAPs)是一种简单的分子标记系统,已经在植物分子作图和基因定位上被证明其有效性[ 9]。该技术是基于正向和反向引物对模板DNA的PCR(polymerase chain reaction,聚合酶链式反应)扩增,引物具有3个明显不同的序列框:5'端存在的10~11个随机碱基序列,紧跟着CCGG(正向引物)或AATT(反向引物)的碱基序列,最后是位于3'端的3个选择性碱基。从技术的可操作性来看,每个引物5'端的10个随机碱基序列可以促进任意序列的扩增,即类似于RAPD(random amplified polymorphic DNA,随机扩增多态性DNA标记)标记的结果[ 10]。正向引物的CCGG 序列可以优先与具有丰富GC碱基的外显子序列退火结合,而反向引物的AATT 序列可优先与具有丰富AT碱基的内含子和启动子序列退火结合[ 9]。引物3'端的3个选择性碱基可以对模板DNA进行选择,与在AFLP(amplified fragment length polymorphism,扩增片段长度多态性)选择性扩增中使用的引物的3'端很相似[ 11]。基于这种独特的引物设计,SRAP标记具有相对于其他标记更好的重复性、稳定性和简易性。SRAP标记已经应用于芸苔属( Brassica)[ 9]、南瓜属( Cucurbita)[ 12]、野牛草( Buchloe dactyloides)[ 13]、紫花苜蓿( Medicago sativa)[ 14]、黑麦草( Lolium)[ 15]和芍药属( Paeonia)[ 16]。Budak等[ 17]发现在揭示遗传关系相近的野牛草品种间的遗传多样性时SRAP标记比SSR(simple sequence repeats,简单重复序列)、ISSR(inter-simple sequence repeat,简单序列重复区间扩增多态性)和 RAPD标记更加有效。
比较具有不同地理分布的植物种质,是进化生物学和植物育种学的重要研究内容。因此,老芒麦地理分布的遗传变异研究,对于收集和管理其种质资源非常重要。然而,目前还未见对不同国家或大的生态区来源的老芒麦种质的遗传多样性水平研究的报道。本研究的目的在于:1)评价SRAP标记在老芒麦种质研究中的有效性;2) 确定来自于3个国家的84份老芒麦种质间的遗传关系;3)检测遗传多样性水平与地理来源的关系。这是首次关于利用SRAP标记检测老芒麦遗传多样性的研究报告。
84份老芒麦供试种质分别从美国国家遗传资源库(NPGS,USDA)、四川农业大学小麦研究所和四川草原科学研究院获得( 表1)。种质的护照信息显示它们分别采集自俄罗斯(西伯利亚地区为主)、蒙古国、中国新疆、中国的青藏高原地区(川西北高原为主)。每份材料取10~20粒种子,置于有盖培养皿内的吸水滤纸上萌发。萌发后的种子转移至内含砂子-泥炭混合基质的盆钵内生长,盆钵置于温室内。植株生长至开花期进行形态学鉴定,标本凭证保存于四川农业大学草业科学系。
| 表1 老芒麦种质材料地理来源 Table 1 List of geographical origin of E. sibiricus germplasm material |
| 续表 1 Continued 1 |
每份种质采集5~10个单株的等量新鲜叶片,用液氮研磨成粉,保存于-80℃低温冰箱内。基因组DNA提取参照Doyle[ 18]描述的CTAB(hexadecyl trimethyl ammonium bromide,十六烷基三甲基溴化铵)法进行。对比已知浓度的标准λDNA与样本DNA在0.8%(w/v)琼脂糖凝胶上的电泳图谱,利用Quantity One 软件(Bio-Rad,USA),计算出老芒麦各种质的DNA浓度。将所有样本的基因组DNA用0.1×TE缓冲液[1 mmol/L Tris-HCl,0.1 mmol/L EDTA(ethylene diamine tetraacetic acid,乙二胺四乙酸),pH 8.0]稀释至10 ng/μL,储存于-80℃冰箱内,供SRAP扩增使用。
引物由上海生工生物工程技术公司合成( 表2)。PCR反应混合液(20 μL总体积)由0.2 mmol/L dNTPs, 0.3 μmol/L正向和反向引物,2.5 mmol/L Mg2+,10×Taq buffer,1 U Taq DNA 聚合酶(北京天根生物技术公司),40 ng 模板DNA组成。扩增在PTC-200 热循环仪内进行(MJ Research,Waltham MA, USA),采用以下PCR程序:94℃初变性5 min;94℃变性1 min,35℃退火1 min,72℃延伸2 min,共5个循环;在随后的30个循环中,退火温度增加至50℃;最后72℃延伸8 min。PCR扩增产物在2%琼脂糖凝胶(含有0.1 mg/mL的溴化乙锭)上用0.5×TBE(tris-borate-edta,三羟甲基氨基甲烷-硼酸-乙二胺四乙酸)缓冲液进行电泳分离,并用BIO-RAD自动凝胶成像系统照相保存。
| 表2 SRAP正向和反向引物序列 Table 2 Sequences of forward and reverse SRAP primers |
只记录清晰可辨的SRAP扩增条带,排除弱带和弥散带。对所有材料,有带(即具有相同分子量)记作1,无带记作0,形成原始二元数据矩阵。
用NTSYS-pc2.11x软件分析二元矩阵,计算各种质间的Nei和Li[ 19]遗传相似系数,利用SAHN模块,基于UPGMA法进行聚类分析,构建聚类树形图。利用NTSYS-pc2.11x软件基于聚类树矩阵计算其协表征矩阵(cophenetic matrix),再利用Mantel检验,计算遗传相似性(GS)矩阵与协表征矩阵间的相关系数,从而反映聚类结果与GS矩阵的符合度[ 20]。利用WINBOOT 软件[ 21]基于bootstrap重抽样法,对聚类树形图节点分支的可靠性进行了检测[ 22],设定重复抽样次数为1000。除聚类分析外,利用NTSYS-pc2.11x软件,基于GS矩阵还进行了主向量分析(PCoA,principal coordinates analysis),特征向量以二维形式显示。PCoA分析可以直观地观察各种质间的相关程度,这是因为二维图上各点之间的距离,反映了各点所代表的各种质之间的遗传相似性。
对每对引物扩增的条带而言,下列参数即:总扩增带数(total number of bands, TNB)、多态性带数(number of polymorphic bands,NPB)、多态性条带百分比 (percentage of polymorphic bands,PP)、多态性信息量(polymorphic information content,PIC)[也可称作杂合度(heterozygosity,H)]、条带信息指数(band informativeness,BI)、分辨力(resolving power,RP)和标记指数(marker index,MI)按下文所述进行计算。 PP=NPBs/TNB, PICi=2 fi(1- fi),式中, PICi 是第 i个标记的 PIC值, fi是扩增带等位基因(即记为“1”的条带)的频率,而(1- fi)是无效带等位基因(即记为“0”的条带)的频率[ 23]。对每个引物组合而言,计算PIC应该求平均值[或称之为平均杂合度(average heterozygosity, Hav),即 PICav=Hav=∑ PICi/N,式中, N是各引物对产生的多态性带数(NPB); BIi=1-(2×|0.5- fi|),式中, BIi是第 i个标记的 BI值, fi是各种质具有的扩增带(即记为“1”的条带)的频率,而 RP=∑ BIi。对每对SRAP引物而言,计算 BI时应该求平均值,即 BIav=∑ BIi/N,式中, N是各引物对产生的多态性带数(NPB)。对每个引物组合而言,标记指数(MI)可以按下述公式计算: MI=NPB×PI
为了解老芒麦种质不同地理类群间和类群内的SRAP变异,利用ARLEQUIN 3.1软件进行了分子变异方差分析(AMOVA)[ 25],计算类群间的遗传分化系数 ΦST。同时利用AMOVA分析,还可以得到不同地理类群间的遗传距离(称之为 Φ统计量)[ 26]。方差成分及类群间遗传距离的显著性检测采用9999次随机置换进行。
从140个引物组合中,筛选出23个可以产生清晰可辨条带的引物组合。这23个引物组合对84份老芒麦种质共产生337条扩增带,分子量范围为75~1580 bp( 图1)。其中,203条(60.24%)为多态性带( 表3)。单个引物组合产生的多态性带为2~19条。平均每个引物组合产生14.7条带,其中多态性带为8.8条。单个引物的PIC值在0.151~0.438之间变化,平均值为0.284( 表3)。引物组合的平均条带信息指数值为0.408,变幅为0.230(Em9+Me15)~0.696(Em12+Me16);23个引物组合的标记分辨力(RP)的变幅为1.119(Em12+Me5)~9.333(Em1+Me10),平均值为3.631。具有较高的分辨力和标记指数的引物组合一般同时具有较高的多态性,能够辨别更多的种质。
| 表3 23对SRAP引物对84份老芒麦种质产生的总扩增带数(TNB)、多态性带数(NPB)、多态性条带百分比(PP)、多态性信息量(PIC)、条带信息指数(BI)、条带分辨力(RP)和标记指数(MI) Table 3 Total number of bands (TNB), number of polymorphic bands (NPB), percent polymorphism (PP), polymorphic information content (PIC), band informativeness (BI), resolving power (RP) and marker index (MI) of the 23 primer combinations used to generate SRAP markers in 84 E. sibiricus accessions |
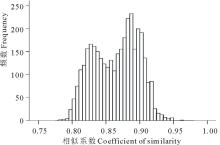
84份种质间的遗传相似性(GS)系数变幅为0.783(204251与PI610886)~0.965(205119与205151),平均值为0.865。 图2描述了3486个GS值的频数分布。前2个最高的GS值频数分别分布在0.886~0.891和0.880~0.885。约95%的GS值分布在0.810~0.921之间,只有0.8%的GS值高于0.93,同时也只有0.1%的GS值低于0.80。这说明供试材料间的遗传关系较近。
基于SRAP标记的UPGMA系统树( 图3), 揭示了84份老芒麦种质间的遗传关系。 系统树可以在不同的GS水平上将供试种质划归为不同的组群。在GS=0.84的水平上,供试种质可以被划分成2个明显不同的组群。第Ⅰ组群包括来自于俄罗斯(20份)、蒙古(16份)和中国新疆(23份)的种质。第Ⅱ组群包括全部来自于青藏高原的25份种质。第Ⅱ组群又可划分为3个亚组:亚组1包括甘肃种质3份、青海种质2份,西藏种质1份;亚组2包括13份四川种质和2份甘肃种质;亚组3包括3份四川种质和1份西藏种质。协表征矩阵与GS矩阵间的相关系数达0.88,说明聚类结果与原始GS矩阵能较好地吻合。基于Bootstrap分析所得的对系统树分支节点置信度的支持值(bootstrap value)被标注在 图3中聚类图上。共有19个分支节点具有高于50%的置信度。第Ⅰ和第Ⅱ组分支节点被91%和100%的bootstrap值支持,表明了青藏高原来源的种质类群(Ⅱ)与其他地理来源的种质类群(Ⅰ)具有明显差异。
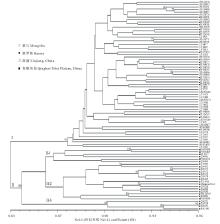 | 图3 基于SRAP数据利用UPGMA法获得的老芒麦种质的聚类树形图Fig.3 Dendrogram of E. sibiricus accessions based on SRAP data using UPGMA method |
基于84份种质GS矩阵的主向量分析(PcoA)的结果表明,第1和第2主向量分别可以解释16.7%和5.8%的遗传变异。依据 图4上各种质间的空间关系,可以直观地看出84份材料被明显划分成两大组,其中青藏高原组又可划分成3个亚组。这与UPGMA聚类结果保持一致。
根据采集地及其生态条件的差异,可以将供试种质划分为四大类群,即青藏高原、新疆、蒙古和俄罗斯类群。基于扩增带(记为1的带)和无效带(记为0的带)的频率,对群体遗传结构进行了分析。AMOVA分析结果显示:在总的遗传变异中有79.62%发生在类群内,有20.38%发生在类群间( ΦST=0.204),群体间和群体内的变异均为极显著( P<0.0001)( 表4)。
| 表4 4个老芒麦地理类群的分子方差分析(AMOVA) Table 4 Analysis of molecular variance (AMOVA) of four germplasm groups of E. sibiricus |
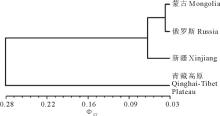
基于AMOVA分析,可以得到类群间两两比较的 ΦST 值,它可以代表标准化的类群间遗传距离(pairwise ΦST distance)( 表5)。青藏高原类群与其他类群间的遗传距离均比较大,而剩余3个类群间的遗传距离比较小。类群间成对 ΦST的变幅为0.0371(蒙古类群与俄罗斯类群间)~0.3547(蒙古类群与青藏高原类群间)。为更好地了解各类群间的遗传关系,利用 表5中的 ΦST距离,进行UPGMA聚类分析( 图5)。4个地理群被明显划分成两支,一支为青藏高原类群,另一支包括蒙古、俄罗斯和新疆类群。青藏高原类群明显区别于其他3个类群,这与对84份种质的UPGMA聚类结果一致( 图3)。
| 表5 4个地理类群间的成对 ΦST遗传距离 Table 5 Pairwise ΦST distances among four ecogeographic groups |
形态-农艺性状和生化标记(同工酶和种子贮藏蛋白)是评价植物种内遗传多样性的常用手段[ 7, 27, 28, 29, 30]。由于不受环境影响、不受限于植物的发育阶段,分子标记目前已成为遗传多样性评价的强有力的主流研究手段。虽然目前有关利用分子标记研究老芒麦种质遗传变异的报道很少,但RAPD、SSR、AFLP、PCR-RFLP等DNA分子标记已广泛用作披碱草属植物的群体遗传结构和种间系统进化研究[ 31, 32, 33, 34]。SRAP标记综合RAPD标记的简便性和AFLP标记的高多态性和稳定性于一身,其上下游通用引物可以搭配使用,极大地提高了引物的利用效率,是一种进行遗传多样性评价、品种鉴定和系统进化研究的有效工具[ 9, 13]。本研究得到的老芒麦种质的SRAP多态性比率(PP)为60.24%,低于已报道的禾本科植物野牛草(95%)[ 13]和鸭茅( Dactylis glomerata)(84.4%)[ 35],而且也低于对青藏高原地区老芒麦SRAP研究的结果(86.48%)[ 36]。本研究利用23个引物组合共扩增出337条带,其中多态性带203条,平均每个引物组合扩增多态性带8.8条,说明SRAP的扩增产率较高,非常适合对大规模种质的遗传变异检测。由于SRAP可以采用更加灵敏的聚丙烯酰胺凝胶银染或荧光标记来检测扩增带,而本实验采用的是较为简便的琼脂糖检测法,这可能是本研究所得的多态性比率较低的部分原因。
84份老芒麦种质间的平均遗传相似系数(GS)为0.865,表明供试材料间具有较近的遗传关系,这很可能与老芒麦的繁育方式相关。一般来说,相对于异花授粉和常异花授粉植物而言,自花授粉植物具有较高的群体间遗传变异和较低的群体内遗传变异[ 37]。由于老芒麦存在较高的异交率,并非严格的自花授粉植物,属于常异花授粉植物,所以每份种质都可以看作是一个内部存在较高变异的小群体,而本实验采用的是混合单株提取DNA法(bulked DNA),可能会掩盖种质内的遗传变异,降低决定种质间遗传关系的多态性带的数目;加上种质间在繁殖后代过程中存在一定的基因流(花粉流)[ 38],这两种因素可能会在一定程度上降低种质间的遗传差异,导致种质间较高平均GS值的出现。根据地理来源的不同,将供试种质划分为四大地理类群。地理隔离、生态隔离和繁殖隔离会极大地影响植物的种内遗传多样性[ 39]。所以青藏高原类群与其他3个类群种质间平均GS值均较小,其原因可能是青藏高原与其他三地区的生态地理环境存在的巨大差异所致。
本研究运用AMOVA分析的结果表明:老芒麦地理类群内的遗传变异占总变异的79.62%,类群间的遗传变异占总变异的20.38%,基于Shannon指数的分析结果与AMOVA分析结果非常接近。另外在4个地理类群中,青藏高原类群具有最高的Shannon多样性(Ho=0.2398)。这表明进行老芒麦遗传资源的挖掘利用时,应更多地重视本地优异资源特别是青藏高原地区资源的收集、保护及隔离繁种。
根据表现所有供试种质间遗传关系的聚类树形图和主向量空间分布图来看,各种质的聚类分布与地理来源有较强的关系[ 40]。种质的地理来源代表了不同的宏观生态环境,而这些不同的生态环境条件是造成种质间的遗传变异的主要因素[ 41]。本研究供试种质被分成两大类的原因,可能主要依赖于青藏高原种质的特有生态地理适应性。由于青藏高原的高海拔及它被喀喇昆仑山、阿尔金山和祁连山等著名高山与其他种质的来源地区隔离开来[ 42],造成其植物种质可能具有特定的生态地理适应性。需要注意的是,来自于新疆、蒙古和俄罗斯的种质并没有按地理来源被清晰的分开,可能与这3个国家或地区接壤且并无明显生态条件差别有关。另外,种质繁殖后代过程中的遗传交换可能会加剧种质间的遗传相似性。同样,基于AMOVA分析产生的类群间 ΦST值的聚类分析也反映了相似的实验结果。虽然AMOVA分析表明地理类群间的变异仅占总变异的20.38%,但达到了极显著水平,这从另一个侧面反映了青藏高原种质类群与其他类群间存在较大的遗传差异。本结果与Chen等[ 43]、陈智华等[ 44]和苗佳敏等[ 45]对垂穗披碱草( Elymus nutans)遗传多样性的研究结果一致,即青藏高原与其他地区的种质材料存在明显的遗传分化。特别提及的是,由于AMOVA可以计算无法估计是否处于Hardy-Weinberg遗传平衡状态或人为划分的植物类群或群体间的 ΦST值作为其遗传距离,因而在评价类似植物群体间和群体内的遗传关系时非常有效[ 46]。如果采用POPGENE等软件强行基于H-W遗传平衡来计算人为划分群体(如地理类群)的遗传分化,可能存在较大误差。
本研究的结果表明SRAP是评价老芒麦种质资源遗传变异的有效手段。今后应增加利用SSR等标记手段以检测非编码区的遗传变异,综合评价现有老芒麦种质在全基因组水平上的遗传多样性,为种质保护、利用和品种选育提供有益的信息。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|
| [30] |
|
| [31] |
|
| [32] |
|
| [33] |
|
| [34] |
|
| [35] |
|
| [36] |
|
| [37] |
|
| [38] |
|
| [39] |
|
| [40] |
|
| [41] |
|
| [42] |
|
| [43] |
|
| [44] |
|
| [45] |
|
| [46] |
|



 s rank correlation coefficient between RAPDs and SSRs was r=0.24 and between ISSRs and SSRs was r=0.66. A genotype-assignment analytical approach might be useful for cultivar identification and property rights protection. Polymorphic SRAPs were abundant and demonstrated genetic diversity among closely related cultivars.
s rank correlation coefficient between RAPDs and SSRs was r=0.24 and between ISSRs and SSRs was r=0.66. A genotype-assignment analytical approach might be useful for cultivar identification and property rights protection. Polymorphic SRAPs were abundant and demonstrated genetic diversity among closely related cultivars. pauciflorum
pauciflorum and
and