谭红妍(1991-),女,山东聊城人,在读硕士。E-mail:hongyantan@yeah.net
土壤微生物群落是土壤质量和生态系统功能变化的敏感指标。为了探讨放牧对土壤微生物群落结构的影响,以呼伦贝尔草甸草原肉牛控制放牧试验为平台,分析4种不同放牧强度(对照区G0.00:0.00 Au/hm2、轻度放牧G0.23:0.23 Au/hm2 、中度放牧G0.46:0.46 Au/hm2 、重度放牧G0.92:0.92 Au/hm2)下温性草甸草原土壤微生物群落结构的变化。结果表明,放牧改变了土壤微生物的组成,但不影响功能菌的优势地位。土壤总微生物量随着放牧强度的增加表现为先减少后增加的趋势,即为对照区最高,中度放牧G0.46最低,重度放牧G0.92出现小幅度的回升;细菌、革兰氏阴性菌、腐生真菌、丛枝菌根真菌(AMF)生物量也呈现相同变化。革兰氏阳性菌、放线菌生物量则随着放牧强度的增加而增加。此外,AMF对于放牧干扰最为敏感(各放牧强度下极显著差异 P<0.01)。除趋势对应分析结果表明,对照区G0.00和轻度放牧G0.23处理下土壤的微生物群落结构较为相似,中度放牧G0.46和重度放牧G0.92处理导致微生物群落结构发生大幅度改变。有机质、速效磷是影响微生物种类和数量的重要养分因素。
Soil microbial community characteristics are an important indicator of soil quality and ecosystem functional changes. In this study, the impacts of cattle grazing intensity on soil microbial community structures were examined by phospholipid fatty acid (PLFA) in temperate meadow steppe on the Hulunber grasslands, northeastern China. Microbial communities under four grazing intensities; 0, 0.23, 0.46 and 0.92 animal units (au)/ha with three replicates were assessed in 2013. The results showed that grazing influenced the composition of the soil microbial community. Total soil microbial biomass was highest under zero grazing, followed by the 0.92 au/ha stocking rate and was lowest under the moderate stocking rate. The biomass of bacteria, Gram-negative bacteria, saprophytic fungi, and Arbuscular mycorrhizal fungi (AMF) responded similarly. There was a large, highly significantly treatment effect on AMF. The biomass of Gram-positive bacteria and Actinomyces trended upward with increased grazing intensity. In addition, we found that soil microbial community structure was similar in the un-grazed and low stocking rate treatments whereas the intermediate and high stocking rates substantial changed the microbial community structure. Soil organic matter and available phosphorus were important factors influencing soil microbial biodiversity and biomass.
土壤微生物群落是草地地下生态系统的重要组成成分, 其不仅是土壤养分重要的“ 源” 和“ 库” , 而且介导许多控制碳氮循环的关键过程, 在草地生态系统过程和功能的维持中扮演着至关重要的角色[1, 2]。微生物生命周期短, 对生存的微环境十分敏感, 土壤微生物群落成为判别干扰条件下土壤生态系统状态的重要生物学指标。脂类物质是构成活体微生物细胞膜的主要成分, 它在细胞中含量稳定约占细胞干重的5%, PLFA即为甲基化土壤中提取磷脂成分后得到的脂肪酸产物[3]。不同的微生物通过不同的代谢途径形成不同的PLFA, 可以作为一种生物标记来鉴定土壤中微生物的种类和丰富度。近年来, PLFA技术以其快速、准确、重现性好、对试验条件要求较低等优点[4], 被广泛用于土壤中微生物群落结构的动态监测。
放牧是草地的主要利用方式之一, 对土壤微生物乃至整个土壤生态系统都有深远影响。适度放牧有助于土壤微生物数量的增加, 过度放牧会导致微生物数量明显减少; 真菌和细菌对放牧强度的响应比放线菌更为敏感[5, 6], 随着放牧强度的增加, 真菌、细菌数量显著下降, 放线菌数量升高。赵帅等[7]发现放牧干扰下土壤微生物的PLFAs种类发生改变, 细菌与真菌PLFAs比值显著升高, 革兰氏阳性菌与阴性菌PLFAs含量比值显著下降。Clegg[8]对英格兰西南一草地研究发现, 放牧可以显著提高革兰氏阳性菌和阴性菌的PLFAs含量, 放线菌与真菌的指示性PLFAs变化不显著。而Ingram等[9]在美国怀俄明州北部的一个半干旱混合草地研究不同放牧强度对微生物群落结构的影响时发现, 不同处理并没有使表征各微生物类群 的PLFAs含量发生显著变化, 在轻度放牧区总PLFAs含量略高。
放牧对地上植被的影响研究的报道较多, 对土壤性状影响的研究也有一些报道, 但大多数的研究内容都局限在土壤三大类群微生物(细菌、真菌、放线菌)的数量、土壤酶活性等研究上。本研究立足于呼伦贝尔草原连续5年的肉牛控制放牧样地, 采用磷脂脂肪酸技术研究不同放牧强度对土壤微生物群落结构的影响, 旨在揭示长期放牧下土壤微生物多样性及群落结构的演变状况, 为深入了解放牧系统地下生态学过程、合理利用草地资源提供依据。
本研究在中国农业科学院呼伦贝尔草地生态系统试验站的肉牛控制放牧样地中进行。研究点位于呼伦贝尔市谢尔塔拉牧场场部东3 km, 属于大兴安岭西麓丘陵向内蒙古高原的过渡区, 地理位置为49° 32'-49° 34' N, 119° 94'-119° 96' E, 海拔670~677 m。研究区属于温带半干旱大陆性气候, 年均温-3~1℃, ≥ 10℃年积温1580~1800℃, 无霜期110 d左右, 年平均降水量350~400 mm, 降水期多集中在7-9月。土壤为暗栗钙土。植被类型为羊草+杂草类, 主要物种有羊草(Leymus chinensis)、裂叶蒿(Artemisia incise)、苔草(Carex tristachva)、贝加尔针茅(Stipa baicalensis)、展枝唐松草(Thalictrum squarrosum)、日荫菅(Carex pediformis)、蓬子菜(Galium verum)、麻花头(Serratula centauroides)等, 伴生种有狭叶柴胡(Bupleurum scorzonerifolium)、斜茎黄芪(Astragalus adsuigens)、山野豌豆(Vicia amoena)、草地早熟禾(Poa pratensis)等。
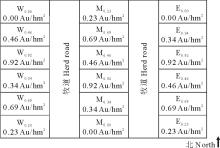
放牧样地共存在6个水平的放牧梯度处理, 载畜率分别为G0.00:0.00 Au/hm2、G0.23:0.23 Au/hm2、G0.34:0.34 Au/hm2、G0.46:0.46 Au/hm2、G0.69:0.69 Au/hm2、G0.92:0.92 Au/hm2。其中以500 kg肉牛为一个标准家畜肉牛单位, 用250~300 kg的放牧肉牛头数来控制不同的放牧强度, 6个放牧梯度肉牛头数分别为0, 2, 3, 4, 6, 8头, 共用69头肉牛。每种处理3个重复, 共包括15个放牧区和3个对照封育区。各处理的分布遵循随机区组试验设计原则, 每个小区为300 m× 160 m, 试验区总面积90 hm2(包括牧道), 具体设计见图1。从2009年开始, 每年6月中旬开始放牧, 10月中旬结束, 为期120 d。整个放牧期肉牛日夜均在放牧区, 肉牛饮水通过拉水供应。
本项研究中选取了4个放牧强度:对照区G0.00、轻度放牧G0.23、中度放牧G0.46、重度放牧G0.92, 野外采样时间为2013年08月15日至19日。每个试验小区随机设置3个采样点, 每个采样点用直径5 cm的土钻取0~20 cm土壤(3点混合), 剔除石块和土壤中的动植物残体后, 采用“ 四分法” 保留约500 g土样。一部分土样带回实验室后立即过2 mm筛保存于-80℃超低温冰箱; 另一部分土样自然风干, 用于测定土壤基本养分。
1.3.1 土壤磷脂脂肪酸提取与分析
将置于超低温冰箱中的土样放入超低温冷冻干燥机中处理后, 取3.00 g左右的冻干土, 利用Blight/Dyer法通过氯仿-甲醇-柠檬酸缓冲液震荡提取总脂, 经SPE柱层析分离得到磷脂脂肪酸; 将得到的磷脂脂肪酸甲酯化。PLFA的定性与定量分析用HP6890气相色谱和Sherlock软件, 以正十九烷脂肪酸甲酯为内标物。各类群微生物标记如表1所示。命名法采用X:Yω Z, 其中X是指主链碳原子个数; Y为双键个数; Z为甲基链离双键的位置。i, a, Me分别表示异构、反异构、甲基支链, cy表示环丙基。
| 表1 估算微生物生物量的脂肪酸 Table 1 PLFA for calculating soil microbial biomass |
1.3.2 土壤基本养分分析
土壤有机质用重铬酸钾容量法— 外加热法进行测定; 全磷采用NaOH熔融-钼锑抗比色法; 全氮采用半微量凯氏定氮法; 土壤pH采用电位法; 速效磷采用0.5 mol/L NaHCO3浸提-钼锑抗比色法; 速效钾采用NH4OAC浸提-火焰光度法; 碱解氮采用1.0 mol/L NaOH碱解扩散法[16]。
微生物群落结构用CANOCO 4.5中的除趋对应分析(detrended correspondence analysis, DCA)并作图。其余数据用SPSS 20.0分析, Excel作图。
本实验共检测出44种磷脂脂肪酸生物标记, 其中G0.23处理下PLFA种类最多, G0.92处理下最少。不同放牧强度下土壤的PLFA种类存在明显差异, 比如15:0 iso 3OH与18:1 2OH为G0.23放牧处理所特有, 16:1ω 9c为G0.46放牧处理所特有。但4个放牧梯度下优势类群并未发生改变, 均为16:00、16:0 10-methyl、18:1ω 7c、19:0 cycloω 8c, 它们含量之和所占比例分别为36.06%, 35.00%, 36.84%, 34.80%。表明不同放牧强度下磷脂脂肪酸组成改变, 即放牧改变了土壤微生物的区系组成和丰富度, 却不会影响优势菌的地位(表2)。
| 表2 不同放牧强度下土壤微生物主要磷脂脂肪酸构成特征(平均值± 标准误) Table 2 The profiles of main PLFAs of microbes in soils from different grazed degrees(Mean± SE)nmol/g |
微生物区系组成中, 细菌比例最高, 其次为放线菌、真菌、AMF。不同放牧强度下, 各大类微生物的特征标记PLFAs含量发生了改变(图2)。土壤总微生物量随着放牧强度的增加表现为先减少后增加的趋势, 即为对照区最高, 中度放牧G0.46最低, 重度放牧G0.92出现小幅度的回升; 细菌、革兰氏阴性菌、腐生真菌、AMF生物量也呈现相同变化。革兰氏阳性菌、放线菌生物量则随着放牧强度的增加而增加(表3)。此外, AMF对于放牧干扰最为敏感(各放牧强度下极显著差异P< 0.01)。
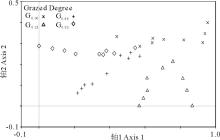 | 图2 不同放牧梯度下土壤微生物群落PLFA的除趋势对应分析Fig.2 Detrended correspondence analysis of PLFA profiles from soil microbialcommunities of different grazed degrees |
| 表3 不同放牧强度下土壤各微生物类群生物量(平均值± 标准误) Table 3 The microbial groups PLFAs content from different grazed degrees(Mean± SE)nmol/g |
为了更直观了解不同放牧强度下土壤微生物群落的分布格局, 对36个土样和主要的磷脂脂肪酸进行排序, 其中前两个排序轴的特征值分别为0.526和0.167, 累积贡献率之和为78.3%。排序图基本上能将4个放牧轻度的样方区分开, 轻度放牧G0.23同对照区G0.00群落结构较为相似, 中度放牧G0.46同重度放牧G0.92群落结构较为相似。表明当放牧干扰超过某一强度时, 会引起土壤微生物群落结构大幅度的变化。
图3为主要磷脂脂肪酸在除趋势对应分析中的因子载荷分析结果。第一排序轴上的载荷值较高的脂肪酸可以分为两类, 表征真菌的脂肪酸(18:2ω 6、16:1ω 5)和表征革兰氏阴性菌的脂肪酸(17:1ω 8、18:1ω 9、18:1ω 7), 它们随着放牧强度的增加而减少。表征革兰氏阳性菌的脂肪酸(a17:0、a15:0、i14:0、i15:0、i16:0)和表征放线菌的脂肪酸(10Me18:0、10Me17:0、10Me16:0)在两排序轴载荷值都较低, 说明这几种脂肪酸在中度放牧G0.46含量最低。
通过对各菌群PLFAs含量与土壤养分相关性分析表明(表4), 土壤总PLFAs含量和细菌、真菌、革兰氏阴性菌、丛枝菌根真菌的PLFAs含量均同有机质、全磷、速效磷、速效钾呈显著正相关(P< 0.05), 尤其是腐生真菌、丛枝菌根真菌的PLFAs与有机质、速效磷相关水平达到了极显著程度(P< 0.01)。由此说明真菌和AMF可用作评价土壤肥力与健康的生物指标, 同时有机质、速效磷是影响微生物数量和种类的重要养分因素。相关分析结果还显示, 革兰氏阳性菌、放线菌PLFAs含量同大多数养分指标成负相关, 各菌群PLFAs含量与全氮、碱解氮、pH间无显著相关性。
| 表4 PLFAs与土壤养分相关性分析 Table 4 Correlation between soil PLFAs content and soil nutrients |
放牧干扰既引起植物生产力的下降, 也降低了植物多样性。生产力下降导致地上的叶凋落物、地下死亡根系的凋落物和根系分泌物的减少, 使得微生物可利用营养物减少[17], 最终引起微生物数量的下降。物种多样性降低会引起植物产物的改变和凋落物有机组分的变化[18, 19, 20, 21, 22], 从而影响微生物群落的组成和功能。与王启兰等[20]、Raiesi和Asadi[22]研究结果一致:随着放牧压力的增大, 土壤总微生物显著下降。放牧显著降低了地上生物量和活根生物量, 微生物分解底物减少。此外, 植被盖度的下降以及牲畜的过度践踏, 引起土壤的含水量、透水性和透气性的下降[20], 土壤微环境遭到破坏, 也会降低微生物的繁殖速率。
不同菌群微生物因自身生长繁殖特性不同, 对放牧干扰存在不同的响应。革兰氏阴性菌与腐生真菌生物量的下降, 干扰的机制可能是:通过改变土壤环境来直接影响微生物生长, 比如有机质含量、养分的有效性[2], 并且家畜活动会严重破坏腐生真菌的菌丝。而在重度放牧G0.92中, 革兰氏阴性菌、真菌、AMF和总微生物量都出现小幅回升, 原因可能在于不稳定基质(尿液与粪便)的输入为微生物提供易利用的营养元素, 适当地促进微生物生长[21]。此外, 本研究中AMF生物量在4个放牧强度下极显著差异(P< 0.01)。Jansa等[23]和Schnoor等[24]曾采用PLFA技术研究不同微生物菌群对外界干扰响应时也发现:AMF对干扰反映最为敏感。AMF与植物是共生关系, 植物物种的变化对其影响最为显著。羊草为该研究区的主要优势种, 也是牲畜较为喜欢的牧草之一。牧压的增加必然会引起羊草优势度的显著下降, 那么与羊草存在共生关系的AMF因菌丝结构的破坏, 其生物量定会显著下降。有研究指出[24], 放牧干扰引起活根生物量的变化是影响微生物群落改变的主要原因, 其中AMF为典型代表。随放牧强度的增加, 革兰氏阳性菌和放线菌生物量呈现出增加的趋势, 同多数文献研究结果不太一致, 有待于进一步验证。Klumpp等[25]在寻找高强度放牧导致土壤碳丢失的原因时, 指出高强度的干扰通过影响植物根系及由它们控制的微生物, 加快了土壤中营养元素的循环。是不是可以有这样一种猜测:高强度放牧使得营养循环效率提高的原因在于, 革兰氏阳性菌和放线菌生物量的增加?
DCA分析结果表明, 过度放牧会引起土壤微生物群落结构的显著改变。本研究中取土深度为0~20 cm, 微生物大部分属于根际微生物[19]。微生物群落能够利用的能源物质主要为根际沉积物(根系分泌物、脱落根细胞、根残体), 不同植物根系沉积物元素组成差异很大[26], 最终引起异养微生物群落结构和功能的变化。当存在外部干扰时, 植物将更多的营养元素分配给地下部分, 根系各种元素的相对含量也会发生变化[27]。有机质、速效磷与微生物菌群极显著正相关, 是微生物生长和活性的主要营养因子, 可能植物本身碳、磷含量是影响微生物种类和数量的生物因素。
放牧改变了土壤微生物的组成, 但不影响功能菌的优势地位。土壤总微生物量随着放牧强度的增加表现为先减少后增加的趋势, 即为对照区最高, 中度放牧最低, 重度放牧出现小幅度的回升; 细菌、革兰氏阴性菌、腐生真菌、AMF生物量也呈现相同变化。革兰氏阳性菌、放线菌生物量则随着放牧强度的增加而增加。此外, AMF对于放牧干扰最为敏感(各放牧强度下极显著差异P< 0.01)。除趋势对应分析结果表明, 对照区和轻度放牧处理下土壤的微生物群落结构较为相似, 中度放牧和重度放牧处理导致微生物群落结构发生大幅度改变。有机质、速效磷是影响微生物种类和数量的重要养分因素。
The authors have declared that no competing interests exist.
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [13] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [27] |
|