作者简介:王芳(1990-),女,山东济宁人,在读硕士。E-mail: wangfang213097056@163.com。高秋(1980-),女,云南保山人,畜牧师。E-mail:gaoqiu1980@gmail.com。
本研究利用15对SSR 引物对161 份高粱属种质资源进行了遗传多样性分析。结果显示,15对引物共扩增出118条谱带,其中88条具有多态性,多态性比率(P)为75.21%;多态性信息含量(PIC值)变幅为0.612~0.806,平均为0.699。161份高粱属材料遗传相似性系数为0.636~0.977,平均基因多样性指数(H)为0.696,Shannon信息指数(I)为1.393。UPGMA聚类分析显示, 161 份高粱属材料在遗传相似系数(GS)为0.682处可分为3组;129份高粱和苏丹草材料在相似系数为0.671处可分为2组。表明SSR标记可以作为高粱属种间以及种内遗传分化分析的有力工具,被分析的高粱属种质材料具有较高的遗传多样性。
Fifteen SSR markers were used to quantify the genetic diversity of the core germplasm collections of sorghum. A total of 161 accessions were assayed. 118 bands were generated, among which 88 (75.21%) were polymorphic. The average value of polymorphic information content (PIC) was 0.699 (0.612-0.806); the average Nei’s gene diversity index (H) was 0.696; and the average Shannon information index (I) was 1.393. The genetic similarities among the 161 accessions ranged from 0.636 to 0.977. The UPGMA dendrogram indicated that the 161 Sorghum germplasm collections were clustered into three groups with a threshold of 0.682, and that the 129 Sorghumsudanense and Sorghumbicolorx× Sorghumsudanense germplasm resources were divided into two clusters at a threshold of 0.671. This study demonstrated that there is considerable genetic diversity conserved in Sorghum germplasm collections and SSR is an ideal molecular marker for assessing this diversity.
高粱属(Sorghum)属于禾本科的须芒草族(Andropogoneae poaceae)[1, 2], 全世界大约有29种, 主要分布于热带到温带地区[3, 4]。我国有5种, 主要分布在东北、西南以及华南地区[3]。高粱属植物多为抗旱作物, 在世界各地被广泛种植, 作为粮食、饲料、工业原料等, 具有很高的经济价值[5]。在我国广泛种植的饲料用高粱属作物有高粱(Sorghumbicolor)、苏丹草(Sorghumsudanense)以及高粱和苏丹草的杂交种高丹草(Sorghumbicolor× Sorghumsudanense), 是国家草种质资源库重点收集的种质资源之一。国家草种质资源库是草种质资源的保存和利用中心, 分析种质资源库所保存高粱属种质资源材料的遗传多样性分布以及遗传背景, 可以丰富入库种质资源的遗传信息, 有助于发掘潜在的优质牧草基因, 为育种提供支持, 还可了解不同来源的高粱属资源的遗传差异, 进而提高资源利用效率。
种质资源遗传多样性分析方法经历了形态、细胞、生化、分子等发展阶段, 目前已有多种分子标记方法被应用到遗传多样性评价中[6, 7], 基于具备多态性高、重复性好、稳定可靠等特点, SSR(simple sequence repeat, 简单重复序列)分子标记技术成为其中比较通用的方法[8]。高粱已经完成了全基因组测序[9], 为开发利用高粱属通用SSR引物提供了便利条件, 近年来在高粱属植物中已筛选得到不少扩增效果较好的多态性引物, 现有的研究主要集中在高丹草和苏丹草一些品系的遗传分析[10, 11, 12, 13], 对高粱属引进以及全国范围内采集的野生、栽培的品种和品系材料的遗传多样性分析尚未见报道。
本研究利用SSR标记对国家草种质资源库保存的161份核心高粱属种质材料进行了遗传多样性分析, 旨在对资源库中高粱属种质资源的多样性分布及遗传背景进行验证和评价, 以期为高粱属种质资源的收集、鉴定、保存及创新利用提供基础信息。
供试材料为国家草种质资源库中保存的所有高粱属材料, 共计161份。其中, 野生材料12份, 品种材料29份, 品系材料120份, 材料编号及来源地信息见表1。
| 表1 供试材料 Table1 Plant materials for this study |
2014年9月将试验材料播种于育苗盘, 每份材料保证出苗数目在100株以上, 置于温室中培养。80%种子的子叶出土2 cm时取样, 每株取0.5 cm左右子叶, 每份样品取100个单株。
本试验采用改良CTAB法[14]提取DNA。将材料在液氮中迅速研磨至粉末状。转移至已加入预热(65℃)好的700 μ L CTAB缓冲液(含10 μ L β -疏基乙醇)的离心管中, 充分摇匀; 置于65℃水浴锅中, 温育1~2 h, 期间每隔15 min摇动一次。加入等体积CI(氯仿∶ 异戊醇=24∶ 1), 摇动混匀, 4℃、12000 r/min离心10 min, 取上清液于离心管, 重复此步; 加入2/3倍体积异丙醇或2倍体积乙醇, -20℃存放1 h; 4℃, 12000 r/min离心10 min, 去上清液; 用70%的乙醇清洗沉淀, 4℃, 12000 r/min离心10 min, 去上清液; 风干, 100 μ L TE溶解。提取的DNA经Nanodrop 2000检测浓度及质量, 置于冰箱-20℃长期保存备用。吸取部分溶液稀释到50 ng/μ L作为工作液。
体系:20 μ L, 其中10× Buffer(含Mg2+)2 μ L, 上游引物(10 μ mol/L)与下游引物(10 μ mol/L)各加0.5 μ L, dNTPs(2.5 mmol/L)0.4 μ L, Taq酶(5 U/μ L)0.1 μ L, 模板DNA(50 ng/μ L)4 μ L, ddH2O 12.5 μ L。引物信息见表2, F为上游引物, R为下游引物, 由生工生物工程(上海)有限公司合成。所用试剂均为天根科技生化公司产品。
| 表2 引物信息及序列 Table2 Primer and sequence information |
程序:94℃预变性5 min; 94℃变性40 s; 退火(退火温度见表2)35 s, 72℃延伸45 s, 35个循环; 72℃延伸5 min, 4℃保存。
变性聚丙烯酰胺凝胶电泳:PCR扩增产物中加入6 μ L Loading buffer (98%甲酰氨, pH 8.0、10 mmol/L EDTA, 0.25%溴酚蓝, 0.25% 二甲苯青), 95℃变性5 min, 冰上冷却, 在6%的变性聚丙烯酰胺凝胶上电泳50 min 左右(80 W 恒功率, 不含预电泳30 min)。银染检测扩增产物。
根据PCR扩增产物在变性聚丙烯酰胺上凝胶电泳的结果, 对相同迁移位置或长度一致的条带进行统计, 有带记为1, 无带记为0, 构成标准的0/1矩阵。利用PIC-CALC 0.6软件计算位点多态性含量(polymorphism information content, PIC)值。采用NTSYS 2.10e软件计算SSR的遗传相似系数(GS), 并利用UPGM法进行聚类分析, 绘制亲缘关系树形图。
利用DataTrans1.0将构成的0/1矩阵数据转换成基因型数据, 再利用POPGENE 1.32软件计算Shannon值(I, Shannon’ s information index)、Nei’ s值(H, Nei’ s gene diversity)、等位基因数(Na, observed number of alleles)与有效等位基因数(Ne, effective number of alleles)。
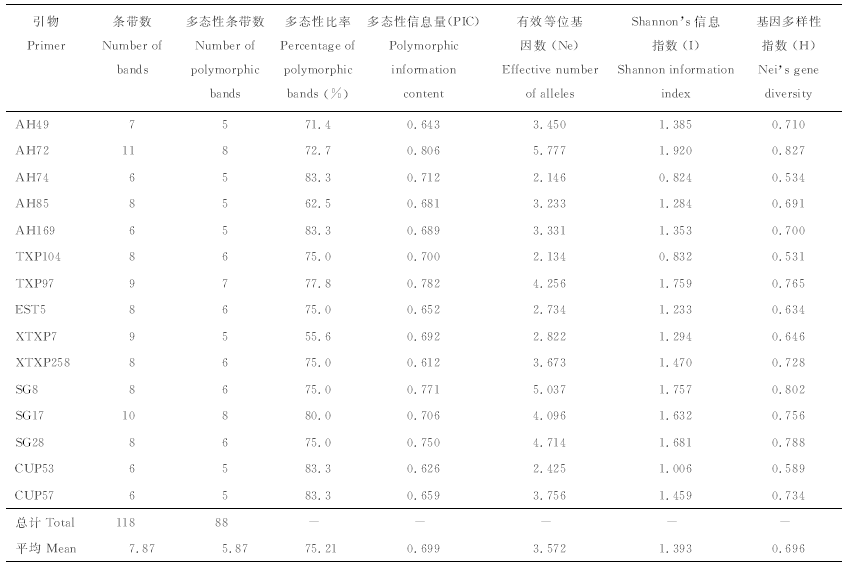
使用已发表文献[11-13, 15]中的108对有多态性的SSR引物进行进一步筛选, 得到15对扩增效果好、多态性高的引物对161份材料进行PCR扩增, 分析结果见表3, 部分材料的扩增图谱见图1。谱带统计结果显示, 15对SSR引物共扩增出118条谱带, 其中多态性谱带有88条, 引物的平均多态性比率(PPB)为75.21%, 各引物扩增谱带数目的变幅为6~11, 平均每对引物扩增出7.87条谱带, 说明所选引物具有丰富的基因多态性。不同引物所揭示的供试材料的多态性信息量(PIC)的变幅为0.612~0.806, 均值为0.699, 161份高粱属材料的基因多样性指数变幅为0.531~0.827, 平均为0.696, 有效等位基因数为2.134~5.777, 平均为3.572, Shannon信息指数为0.824~1.920, 平均为1.393, 上述统计分析结果表明供试材料遗传分化丰富。
| 表3 引物多态性信息 Table3 The polymorphism of SSR primers |
根据扩增结果统计谱带, 计算相似性系数和遗传距离。结果显示161份供试材料的遗传相似性系数为0.341, 变幅为0.636~0.977。其中, 遗传相似系数最大的是37和38, 达到0.977, 其次是26与27、19与21、58与60、106与107、126与134, 达到0.955, 表明37与38、26与27、19与21、58与60、106与107、126与134亲缘关系很近。登记信息显示, 37来源于北京, 38来源于美国, 均为高丹草品种乐食; 26来源于甘肃, 27来源于新疆, 均为苏丹草品种新苏2号。登记信息与遗传相似性分析的结果高度一致, 不同来源地的同一品种材料聚在了一起, 证明所选用引物能够很好地揭示种质材料之间的亲缘关系。其他聚在一起的材料由于登记信息不完善, 需要开展进一步的田间试验进行验证。
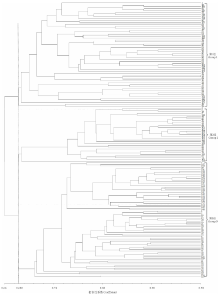
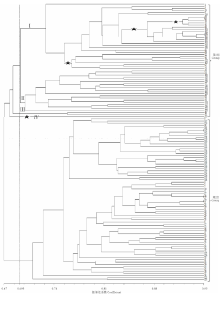
基于遗传相似性系数, 利用UPGMA法构建了161份高粱属材料的聚类分析图(图2)以及129份高粱和苏丹草的聚类分析图(图3)。
在相似性系数为0.682的水平上, 161份材料被分为3组, 第1组主要为高丹草以及进口高粱, 第2组主要为国内的高粱, 第3组主要为苏丹草。其中, 编号为123的高粱聚在了苏丹草一类; 编号为124的苏丹草聚在了高丹草以及进口高粱一类。经核查, 123和124这两份材料为同一单位同一批次上交的种子, 通过对两份材料的种子形态观察以及与材料上交单位核对, 证实材料高粱123应为苏丹草、苏丹草124应为高丹草, 可能是由于登记、复检或干燥过程中的失误导致的。此外, 编号为40的高丹草和编号为3的高粱聚在了苏丹草一支中; 编号为22的苏丹草聚在国内高粱一支中, 以上3份材料登记信息不完善, 还需通过田间试验进行验证。高丹草是以高粱为母本、苏丹草为父本育成的材料, 从聚类图上看其亲缘关系更接近高粱。
在129份高粱和苏丹草的聚类分析中, 在相似性系数为0.671的水平上, 供试材料聚成两大组, 第1组所有材料均为高粱(除了存疑材料22), 第2组所有材料均为苏丹草(除了存疑材料3), 说明我们选用的SSR引物可较好的将高粱与苏丹草区分开。根据种子入库登记的信息, 种质资源库中的高粱种质资源来源广泛, 国内来源有东北(吉林、辽宁)、华北(北京、河北、内蒙古)、华东(山东、江苏)、华中(河南、江西)、西北(宁夏、新疆、甘肃)、西南(重庆、四川), 同时还收集有不少国外高粱材料。从聚类图上也可看出高粱种质资源地域性强, 在高粱这一组中, 在相似性系数为0.693的水平上, 55份材料可分为4个亚组(I、II、III、IV), 大部分材料(52份)聚在I组和II组中, 其中II组主要为来源于东北和国外的高粱材料, 来源于国内其他5个分布区的材料主要聚在I组。在I组中, 大部分来源于华东、华中和西北的材料也聚成了不同的小支。聚类结果显示所分析的高粱种质资源来源较为丰富, 具有较强的地域性。苏丹草种质资源来源比较单一, 主要来自甘肃、宁夏、内蒙古, 在聚类图中并没有表现出很强的地域性。
SSR分子标记技术自20世纪90年代出现以来, 被各国研究者选用进行多个领域的研究和应用[16, 17]。在21世纪初以前由于其高昂的开发成本和实验技术的不完善, 其优越性没有得到完全发挥, 近十年来DNA测序技术的飞速发展, 在很大程度上解决了SSR分子标记技术遇到的瓶颈问题, 因此SSR技术得到了空前的应用和发展[18, 19, 20]。SSR位点具有丰富的多态性, 在品种间甚至于个体间都有变异, 适用于进行种内遗传多样性分析。在种间水平上, 由于变异位点两端保守序列的变异太大, 导致一般无法设计通用引物进行分析, 限制了其在种间遗传多样性分析的应用[21]。本研究利用根据高粱基因组序列设计的SSR引物, 对高粱及其近缘种高丹草和苏丹草进行了分析, 结果表明高粱的SSR引物在高丹草和苏丹草中具有很好的通用性, 可以用来进行这3种材料的遗传多样性分析, 说明SSR引物不限于种内材料的分析, 但在多大范围内可以通用还需要对高粱属其他材料以及高粱属近缘属种材料进行分析。詹秋文等[15]使用RAPD标记对高粱属的两个物种进行了分析, 无法区分高丹草和苏丹草, 我们选用的SSR引物能够区分开高粱和苏丹草两个物种, 在一定程度上说明SSR分子标记在种间水平的应用可能还有很大的空间。
草种质资源是进行草品种改良和种质创新的基础, 也是环境保护和生态建设的重要载体[22], 草种质资源的遗传多样性分析是开发利用优良基因资源、保护基因多样性的有效手段。全国畜牧总站国家草种质资源库目前已保存草种质材料3.2万份, 本研究分析的161份高粱属材料均来源于该库。利用SSR分子标记技术对国家草种质资源库的种质材料进行遗传多样性分析, 一是有助于保证种质资源信息的准确性, 提高入库种质资源的权威性。例如我们通过分析发现登记为高粱的123号材料和苏丹草材料聚在一起, 登记为苏丹草的124号材料和高粱材料聚在一起, 经验证是登记失误造成的。还有一些材料没有聚在合适的位置上, 很有可能也是登记错误造成的, 需要进一步进行验证, 以确保种质资源信息的准确性。二是利用遗传多样性分析可对种质材料的亲缘关系进行梳理, 为培育新品系提供基础信息, 提高种质材料的利用率。目前杂交育种是主流育种方式, 育种材料的亲缘关系是杂交育种工作的重要指导依据, 明晰材料的遗传背景能够大大提高育种的成功率。例如遗传多样性分析验证了编号为26和27以及编号为37和38的材料分别为来源地不同的同一品种材料, 表明通过SSR标记分析能够准确识别高粱属种质资源的遗传距离, 提高种质资源创新利用效率。此外, 遗传多样性分析能够为重点收集提供科学的指导信息。目前草种质资源收集的重点已经从广泛收集转为重点收集, 通过对高粱属种质材料的多样性进行分析, 明晰了高丹草和苏丹草来源比较单一, 多样性不够高的现状, 可为下一步高粱属种质资源的重点搜集、有效保护工作提供科学指导, 有利于进一步明确收集保护的方向。
The authors have declared that no competing interests exist.
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [15] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [22] |
|