作者简介:韦兴燚(1992-),男,福建宁德人,在读硕士。E-mail: weixy16@lzu.edu.cn
在漫长的进化过程中,高等真核生物从原核生物中获取了大量基因,对这些基因进行鉴定分析,可为研究高等植物系统演化及基因组水平比较分析提供理论依据。为了进一步解析大豆和百脉根古老原核基因在其基因组中所起的作用及进化关系,本研究利用生物信息学技术对大豆和百脉根基因组古老原核基因进行全基因组鉴定,并对其特征及功能进行了比较分析。研究结果表明,大豆(40.6%)古老原核基因的占比高于百脉根(33.9%),且多分布于内共生细胞器线粒体、叶绿体中。此外,古老原核蛋白质的结构域在大豆和百脉根基因组中的分布相似,表明其在大豆和百脉根进化过程中具有较高的共线性和保守性。通过GO注释发现大豆与百脉根中古老原核蛋白主要分布在膜系统、细胞、细胞组分中。在分子功能上,大豆古老原核蛋白更多地参与代谢和发育过程,这可能与其接受了更多的人为选育有关。在生物过程中,古老原核蛋白更多参与催化反应和结合反应,主要以酶的形式在发挥作用。通过比较分析,本研究揭示了古老原核蛋白在大豆和百脉根中进化的独特模式,可为其他豆科植物基因组分析提供一定理论基础。
Eukaryotes have acquired a large number of genes from prokaryotes during their evolution. The identification and analysis of these genes can provide a theoretical basis for study of the systematic evolution of higher plants. In order to further investigate the significance old prokaryotic genes during the evolution of Glycine max and Lotus corniculatus, the old prokaryotic genes in these two species’ genomes were comprehensively identified using bioinformatics methods and the functions and relations of these genes were further analyzed. Our results suggest that the number of old prokaryotic genes is higher in G. max (40.6%) than in L. corniculatus (33.9%). The largest fractions of these protein-encoding genes were found to be localized in endo-symbiotic organelles, such as mitochondria and chloroplasts.Similar distributions of the domain architecture of old prokaryotic proteins were found in both G. max and L. corniculatus genomes, revealing that they may share high collinearity and conservation during evolution.Based on Gene Ontology (GO) functional classifications, we found that the old prokaryotic proteins were mainly distributed in membrane, cell, and cell parts. In terms of molecular function, the old prokaryotic proteins mainly participated in metabolic and developmental processes in G. max, which may have been caused by human selection pressure for better agronomic productivity.In terms of biological processes, the old prokaryotic proteins mainly participated in catalytic reactions and binding reactions, indicating that they may play a role as enzymes. Using comparative analysis, our work has indicated a distinctive pattern in the evolution of old prokaryotic proteins in G. max and L. corniculatus, with the results suggesting a theoretical basis for the genomic analysis of other leguminous plants.
进化是指种群在不同世代间通过基因复制、横向迁移、融合以及分裂等多种事件导致其遗传性状发生变化的过程。基因是遗传物质的载体, 研究发现, 具有新功能基因的产生可进一步影响物种的生长发育及对外界环境的响应过程[1, 2]。在众多进化事件中, 从其他基因组中获得基因是真核生物进化的一种普遍方式, 且已在真核生物线粒体、叶绿体的研究结果中得到证实[3]。因此, 真核生物的进化也被认为是伴随着大量真核生物基因迁移的结果[4]。真核生物的基因来源较广, 其基因组具有较高的异质性, 不同基因组间的基因不仅存在功能差异, 系统进化过程也各不相同, 这为鉴定基因的进化年龄提供了条件。研究表明, 如果某些基因在不同物种中存在其直系同源基因, 则可被认为是进化年龄较长的古老基因。相反, 一些基因由于突变、替换等事件的发生而在其他物种中不存在其直系同源基因, 则被定义为进化年龄较短的新基因[5, 6, 7]。在生物进化过程中, 真核细胞从原核细胞中获得大量古老基因, 这些基因在植物生长发育以及响应逆境胁迫过程中发挥着重要作用[8, 9]。通过比较基因组学技术对不同物种基因的比较分析是了解不同物种基因组进化强有力的工具。研究发现, 即使是遗传关系非常相近的物种, 其基因组也存在着显著差异[2]。因此, 鉴定和分析不同物种基因组中的新、老基因差异及分布特点, 可为解析真核生物进化与基因功能研究提供一定的理论依据。
豆科为被子植物中仅次于菊科及兰科的3个最大的科之一, 也是人类利用最为广泛的植物科类之一。大多数豆科植物具有根系庞大、适生范围广、抗逆性强、耐瘠薄、易栽培、生长迅速、萌蘖力强、生物量高、热值高等优点。另外, 豆科植物根部具根瘤, 能固定和利用大气中的游离氮, 对作物发育和植被的建立都有重要意义[10, 11]。豆科植物含有大量的蛋白质、矿物质和丰富的维生素, 具有很高的饲用价值。此外, 部分豆科植物可供人类食用、入药、用于化工业生产等; 有些豆科植物还具有较高的观赏价值, 可应用于旅游景区发展旅游业[12]。其中, 大豆(Glycine max)是世界上重要的粮食和油料作物之一, 是人类食物中植物蛋白质和油脂的主要来源, 与人们的日常生活息息相关。由于其营养价值很高, 也被称为豆中之王、田中之肉。百脉根(Lotus corniculatus)的再生性好, 遗传转化效率相对较高, 是豆科生物学和基因组学研究的模式植物[13]。随着基因组测序的发展, 大豆与百脉根全基因测序已经完成, 为这两种豆科植物功能基因发掘、比较基因组学分析以及系统进化研究奠定了基础[14]。目前, 仅有研究对拟南芥(Arabidopsis)与水稻(Oryza sativa)进行了全基因组古老原核基因的鉴定比对[2], 尚未见有关豆科植物古老原核基因的全基因组鉴定和比较分析。本研究通过对大豆和百脉根全基因组的鉴定与比较得到古老原核基因在植物中的作用, 并揭示这些基因的进化方式, 拟为豆科植物系统进化研究提供相关理论基础。
大豆和百脉根基因组、蛋白质组数据分别来自于Phytozome v11.0 (https://phytozome.jgi.doe.gov/pz/portal.html)和百脉根测序基因组数据库(http://www.kazusa.or.jp/lotus/index.html)。真核生物蛋白质序列来源于COGs数据库(http://www.ncbi.nlm.nih.gov/COG/)。细菌蛋白质序列来源于NCBI数据库(ftp://ftp.ncbi.nlm.nih.gov/genomes/refseq/bacteria/)。利用CD-HIT在线工具[15]去冗杂得到58750658条细菌蛋白质序列用于后续分析。
为了鉴定大豆和百脉根中的古老和新基因, 利用本地BLASTP方法(E-value< 10-6), 分别将大豆、百脉根蛋白质序列与58750658条细菌蛋白质序列进行比对分析。若豆科植物蛋白在细菌蛋白中存在直系同源物则该蛋白为古老原核蛋白, 否则该蛋白为新蛋白。将这些豆科植物中的古老原核蛋白命名为古老原核蛋白, 非古老原核蛋白则命名为新蛋白[7]。利用MCScanX软件分析大豆与百脉根基因间的共线性(http://chibba.pgml.uga.edu/mcscan2/), 选择BLASTP期望值(E-value)1e-10作为阈值。
利用本地PfamScan软件对古老原核蛋白结构域类型进行预测分析[16]。利用在线工具MEME(multiple expectation maximization for motif eliciattion)对获得的大豆、百脉根古老原核蛋白进行结构域预测。通过PfamScan软件对两物种的保守结构域、家族、模体、重复序列进行鉴定。以上分析都采用默认参数设定。
将经过去冗余的基因序列通过Blastx方法与NCBI无冗余Nr数据库进行比对, 分别获得大豆与百脉根每条古老原核基因的GO注释, 再利用WEGO在线软件[17]读取所有的序列, 并对获得的GO注释进行分类。
将大豆与百脉根的蛋白质序列在细菌蛋白质序列中进行BLASTP搜索[18], 分别鉴定并定位出这两种豆科植物线粒体、叶绿体及细胞核内每条染色体中的新、古老基因的数量(图1)。在大豆中共鉴定出87144对基因, 其中新、古老基因数量分别为51770和35374个, 占比分别为59.4%和40.6%。在百脉根中共鉴定出20689对基因, 其中新、古老基因数量分别为13672和7017个, 占比分别为66.1%和33.9%。从中可发现, 不管是大豆还是百脉根, 其基因组中的古老基因数量都小于新基因数量, 说明其在进化过程中产生了大量的新基因。利用MCScanX软件对大豆和百脉根共线性分析发现, 25497个大豆蛋白与13553个百脉根蛋白之间具有共线性关系, 分别占大豆和百脉根蛋白总数的29.26%和65.51%。此外, 发现大豆的第19号染色体和叶绿体与其他染色体及线粒体不同, 其古老基因数量大于新基因数量。在百脉根中, 除线粒体和叶绿体外, 所有染色体中都是新基因数量大于古老基因数量。
对大豆和百脉根基因组中古老原核基因的分析发现, 两物种基因组中古老原核基因所占百分数大豆相对高于百脉根(图1)。在大豆基因组中, 40.6%的基因是古老原核蛋白的编码基因, 而在百脉根中其占比为33.9%。古老基因在每条大豆染色体上的数量百分比为37.7%~52.4%, 数量最低的是第4号染色体, 第19号染色体拥有的这类基因的数量是最多的, 也是大豆染色体中唯一古老基因数量高于新基因的。在百脉根中, 第3号染色体古老基因的数目最小(32.0%), 而在第4号染色体中为最大值(36.2%)。在叶绿体中, 古老基因在大豆和百脉根叶绿体总基因中占比分别高达89.2%和89.2%。而在线粒体中, 古老基因在两物种线粒体总基因中占比分别为34.1%和85.3%。在大豆和百脉根的线粒体和叶绿体中古老原核基因比例较高才符合内共生起源理论。但在大豆线粒体中, 古老原核基因占比却小于真核基因。从数据中发现, 古老原核基因在数量上不集中在两个基因组中的任何特定的染色体上, 仅在大豆第19号染色体中分布较突出。
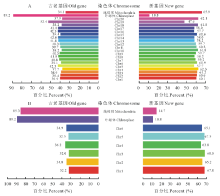 | 图1 大豆(A)与百脉根(B)新老基因染色体分布Fig.1 The distribution of old and new prokaryotic genes on various chromosomes in G. max (A) and L. corniculatus (B) |
真核生物蛋白质的结构域被认为是一种独立的进化单位[19]。通过PfamScan本地软件搜索从PFAM数据库中得到的结构域数据, 所得蛋白质中古老与新蛋白中结构域类型及其所占百分比结果见图2。被归类为新、古老的蛋白质数量分别为35514/53133(大豆)和6719/8132(百脉根)。此外, 不管是大豆的两种蛋白还是百脉根的两种蛋白, 拥有越多的结构域的蛋白质往往数量会越少, 且只有1个结构域的蛋白质数量都占蛋白质总数的1/2以上。另外, 大豆中有1或多个结构域的蛋白质中古老原核蛋白的占比都高于50%, 而百脉根中拥有8个结构域的蛋白质其古老原核蛋白占比低于50%, 仅为47.1%。大豆中古老原核蛋白在拥有10及以上数量结构域的蛋白质中占比最高, 其值为63.4%, 而在拥有2个结构域的蛋白质中占比最低, 其值为55.9%。百脉根中古老原核蛋白分别在拥有6个结构域和8个结构域的蛋白质中占比最高(83.3%)与最低(47.1%)。通过统计不同结构域在大豆和百脉根蛋白中出现的次数发现, 无论是在大豆还是在百脉根中, 新和古老原核蛋白中出现次数最多的结构域都分别为PPR和Pkinase。另外, PPR-2, WD-40, myb-DNA-binding结构域也同时在两种豆科植物的新、古老原核蛋白中出现次数较多。为了验证两物种蛋白质结构域的分布情况, 通过对两个物种的结构域分布进行双样本Kolmogo rov-Smirnov检验[20] (P≤ 0.05), 检验结果表明, 两物种结构域分布无显著差异(P> 0.05), 即两物种古老原核蛋白结构域分布相似。经过进一步检验发现, 该相似性适用于所有的组合, 比如古老原核蛋白(大豆和百脉根), 新蛋白(大豆和百脉根), 大豆(原核和新蛋白), 百脉根(原核和新蛋白)。这些结果表明大豆和百脉根蛋白质结构域具有一定保守性。
大多数基因在不同生物中的同源基因拥有相同的主要生物学功能, 因此, 在某些物种里已知的基因功能信息可以用来解释其他物种对应的同源基因[21]。本研究中, 通过蛋白家族数据库(http://pfam.janelia.org/search/sequence)提供的GOSlim分配进行GO注释, 在大豆中得到2482个GO号, 共注释了26210个基因, 百脉根中则得到14608个GO号, 注释了11964个基因。并将大豆与百脉根中确定的古老原核蛋白质分类为三大GOSlim类别, 即细胞成分、分子功能和生物过程。
2.3.1 细胞成分 大豆中古老原核蛋白在膜系统(membrane)、细胞组分(cell part)、细胞(cell)3个位点上所占比例最大, 达到了29.2%、17.5%、17.5%, 百脉根中的古老原核蛋白同样在细胞组分及细胞方面分布最多(占比皆为18.1%), 但其在膜系统上的分布不如大豆明显, 仅占11.5%。除此以外, 大豆古老原核蛋白还在膜部分(membrane part, 13.9%)、细胞器(organelle, 9.5%)、大分子复合物(macromolecular complex, 8.6%)中分布较多, 百脉根古老原核蛋白则在细胞器(organelle, 14.6%)、细胞器部分(organelle part, 9.2%)、膜部分(membrane part, 7.1%)中分布较多。由两物种中古老原核蛋白的分布可看出其遍布在植物细胞中, 广泛参与了细胞生命活动。
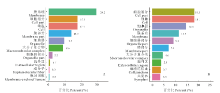 | 图3 大豆(A)与百脉根(B)古老原核蛋白细胞成分Fig.3 The cellular component of old prokaryotic proteins in G. max (A) and L. corniculatus (B) |
2.3.2 分子功能 古老原核蛋白质被发现参与各种结合过程, 如DNA和RNA结合、蛋白质结合、核苷酸结合等。从图4可以明显看出, 大豆与百脉根中的古老原核蛋白在催化活性(catalytic activity)及结合反应(binding)中两者所占比例之和达到了80%左右, 可见其起着非常重要的作用, 这与前人研究结果相符[2]。除此以外, 两物种中的古老原核蛋白都参与了转运过程(transporter activity), 结合前面的催化与结合可推测古老原核蛋白可能是以酶的形式参与这些反应, 而这些反应具有较高的保守性, 因此这些古老原核蛋白被大量的保存了下来。
2.3.3 生物过程 从图5可以发现, 古老原核蛋白质主要参与了代谢过程。大豆的古老原核蛋白在新陈代谢过程(metabolic process)、细胞过程(cellular process)、单生物代谢过程(single-organism process)中所占比例最大, 分别为33.0%、27.7%、18.1%。而百脉根的古老原核蛋白则功能众多且较分散, 但其蛋白所起功能占比最大的3个方面仍与大豆相同。大豆与百脉根的这点差异可能是因为大豆作为重要的经济作物被较多进行人为选择所致。大豆与百脉根在对逆境的响应上差异较大(大豆为3.9%, 而百脉根为9.1%), 这说明在百脉根中有较多的古老原核蛋白质参与逆境生理响应过程。除此以外, 大豆与百脉根古老原核蛋白都在定位(localization)、生物调节(biological regulation)中起着一定的作用。
为了明晰蛋白质在各种不同复杂过程中可能所起的作用, 往往需要深入挖掘相关的基因组[22]。众所周知, 大豆是世界上重要的粮食与油料作物, 其基因组具有极高研究价值。而百脉根是豆科植物与基因组学中的模式植物, 因此通过对这两种植物基因组进行比对研究可为两物种蛋白质研究提供一定理论基础。古老原核基因包含着许多远古时期的功能组件, 如今则形成了现代植物基因组的一个重要部分。早期, 内共生演化导致细胞从内共生细胞器中获得众多基因, 这些内共生细胞器后来演变成叶绿体和线粒体[23, 24]。此外, 还有大量内共生基因后来被转移到宿主的细胞核[23, 25, 26, 27]。从古老原核基因的全基因组鉴定所得的结果也同样证实, 古老原核蛋白质编码基因不仅仅大量存在于叶绿体和线粒体, 也同样大量存在于大豆和百脉根的核基因组(图1)。类似的结果同样也存在于拟南芥(Arabidopsis thaliana)和水稻(Oryza sativa)两种模式植物中[2]。早期的研究也证明, 快速进化的基因可能会由于基因的进化速率和年龄之间的反比关系而被归类为年龄较短的新基因[28]。通过鉴定, 大豆拥有更多的古老原核基因, 这表明百脉根中快速进化的基因数量大于大豆。
前人对拟南芥和水稻两种模式植物的古老基因的鉴定分析中发现, 这两种模式植物线粒体中古老基因的数目都多于新基因的数目[2]。本研究也在百脉根中发现类似情况(图1)。但在大豆中却表现为新基因数量多于古老基因(图1), 这可能与线粒体的功能以及大豆被人为筛选进化有关。线粒体是真核细胞重要的细胞器, 其在细胞能量代谢、生物合成和细胞死亡(包括细胞凋亡和细胞程序性坏死)的调控中起关键作用[29, 30]。有研究显示多种经济作物如甘蓝型油菜(Brassica napus)、水稻等都有线粒体中的基因转入核基因组的情况发生, 这被认为与其功能和进化有关[31]。但另有研究发现植物线粒体基因组在不断地扩大且越来越复杂[32], 因此线粒体中的新基因会越来越多。此外, 可能由于人为筛选高产高质大豆的原因, 线粒体作为能量工厂也被不断筛选, 导致大豆线粒体中的基因不断进化, 新基因数量便大量增加。由于百脉根不论在利用程度还是使用广度都远不及大豆, 因此被人为干预较少, 基因组变化也小于大豆。
结构域是能折叠形成稳定核心的独立单位, 也是蛋白质结构、功能和进化的独立单位[33, 34]。结构域的结合和重组会导致具有新功能蛋白质的形成[35]。因此对结构域的分析可揭示不同结构域及两个物种之间的进化关系。前人研究认为, 在植物中结构域的结构具有较高的相似性, 该结论适用于不同基因组的所有类型的蛋白质结构域[36]。本研究发现, 两物种的结构域具有一定保守性, 不仅分布相似, 而且古老原核蛋白占比也普遍较高(图2)。
原核生物是指由无核膜包被的细胞核和其他的生物膜细胞器组成的生物。因此, 大量的古老原核基因可能位于内共生细胞器, 特别是叶绿体和线粒体中。之前的研究也证实, 在拟南芥和水稻中, 古老原核蛋白都主要定位于叶绿体、膜系统中[2]。本研究的GO注释结果也显示, 大豆与百脉根中的古老基因在膜系统、细胞组分、细胞3个位点上所占比例最大(图3), 与其他研究相类似[37]。此外, 对蛋白分子功能GO注释结果表明, 古老原核蛋白主要具有酶催化功能及结合功能, 这说明它们在基因的调节作用以及应对各种环境条件改变地响应时具有重要作用。类似的结果也存在于拟南芥和水稻中[2]。而在蛋白生物过程GO注释的结果中, 大豆中古老原核蛋白主要参与新陈代谢过程、细胞过程, 而百脉根中的古老原核蛋白质较平均地参与多个方面的过程, 表明大豆受人为选择较多, 导致其蛋白在特定功能上表现突出。
在大豆和百脉根中, 古老原核基因大量位于内共生细胞器如叶绿体、线粒体等, 且这些基因编码的蛋白多位于各种膜结合区域以及参与各种结合功能, 如核苷酸和DNA、RNA的结合, 揭示了其在各种作用调控基因表达的过程中具有重要作用。在大豆中, 古老原核蛋白参与蛋白质代谢和其他发育过程; 而在百脉根中, 这些蛋白质在相关方面的作用相对较弱。因此, 大豆古老原核蛋白质可能由于选择压力使得自身产生较大特异化。此外, 两物种结构域分布相似表明其结构域具有一定保守性, 但这些古老原核蛋白质在功能方面具有多样性。
The authors have declared that no competing interests exist.
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|
| [30] |
|
| [31] |
|
| [32] |
|
| [33] |
|
| [34] |
|
| [35] |
|
| [36] |
|
| [37] |
|





