作者简介:康文娟(1990-),女,甘肃兰州人,在读博士。E-mail: 1103683808@qq.com
根据表型和共生效应,对种内根瘤菌划分生物型,为苜蓿与根瘤菌高效共生育种提供理论依据。以分离自甘肃省3个栽培区域5个苜蓿品种植株不同部位的根瘤菌株为材料,进行16S rRNA测序、多位点序列分型(Multilocus Sequence Typing, MLST)、表型数值分类和共生效应测定。结果表明,78株初筛菌经16S rRNA和MLST分析有20株属于 Rhizobium radiobacter;表型特征数值分类将其聚为7个群。接种5个苜蓿品种,选择对共生效率贡献率最大的地上干重进行多重比较。当接种处理地上干重显著高于未接种处理CK时标记共生效应为A,与CK差异不显著时标记为B,显著低于CK时标记为C。以苜蓿品种为甘农3号、甘农9号、陇中、清水和WL168HQ的顺序,将各菌株共生效应结合,形成6种共生模式。结合表型和共生模式,20株 R. radiobacter被划分为14种生物型,WL168HQ、甘农3号和陇中苜蓿品种内生物型多样性丰富,甘农9号和清水苜蓿品种内生物型单一。综上所述,根据表型特征和共生模型可以进行种内根瘤菌株的生物型划分,苜蓿品种内根瘤菌生物型多样性丰富。
This study aimed to classify biotypes in the same rhizobium species according to their phenotypic characteristics and the symbiotic efficiency between the alfalfa variety and rhizobium strain, and so to provide a knowledge base for breeding rhizobia that are effective symbiotic partners with alfalfa. Rhizobium strains were isolated from different plant parts of five Medicago sativa varieties in three cultivation regions of Gansu province. 16S rRNA sequencing, multilocus sequence typing (MLST), numerical taxonomy analysis based on phenotypic characteristics and symbiotic efficiency determination were used to achieve rhizobia biotype classification. Of 78 rhizobia strains in a preliminary screening, 20 were assigned to Rhizobium radiobacter by 16S rRNA sequencing and MLST analysis. Seven phenotypic clusters were formed based on numerical taxonomy analysis of phenotypic characteristics. Seedlings of the five M. sativa varieties were inoculated with the 20 R. radiobacter strains, and shoot dry weights, which contributed the most to the symbiotic efficiency between alfalfa and rhizobium, for varieties inoculated with each strain were compared using Duncan’s multiple range test. The symbiotic efficiency of strains was designated A, B, or C when the shoot dry weight of inoculated plants was, respectively, significantly higher than, not significantly different from, or significantly lower than that of non-inoculated control plants.The designated symbiotic efficiency of each rhizobia was combined according to the order of M. sativa cv. Gannong No.3, Gannong No.9, Longzhong, Qingshui, and WL168HQ, and six kinds of symbiotic combination patterns were formed.The 20 R. radiobacter strains were divided into 14 biotypes according to phenotypes and symbiotic patterns. For M. sativa cv. WL168HQ, Gannong No.3, and Longzhong many rhizobium biotypes were found, while the rhizobia of M. sativa cv. Gannong No.9, and Qingshui were monotypic. In conclusion, rhizobia of the same species ( R. radiobacter) could be classified into biotypes using information on phenotypic characteristics and symbiotic patterns, and some alfalfa varieties were found to host a number of rhizobium biotypes.
紫花苜蓿(Medicago sativa)是重要的豆科牧草, 具有很强的环境适应能力和丰富的粗蛋白含量[1]。根瘤菌是有益的土壤细菌, 能够与苜蓿共生结瘤固氮, 促进苜蓿生长[2, 3, 4]和增强土壤肥力[5]。根瘤菌亦能够在植物组织内定殖, 增强植株的生物[6]和非生物[7]抗性。目前根瘤菌的分类倾向于在种水平上进行[8], 常用的微生物分型技术又在相对分辨率、时间和成本上存在差异[9], 重复性、再现性低。研究发现持家基因(housekeeping gene)在各个发育阶段所有组织中均恒定表达, 是维持细胞基本生命活动所必需的一类基因。持家基因多位点序列分型(multilocus sequence typing, MLST)通过直接比较核苷酸序列鉴定了至少7个位点的等位基因[10], 相比微生物常用分型技术有重大改善。Berkum等[11]应用MLST技术对土壤根瘤菌种群基因结构进行分析, 鉴定出与紫花苜蓿结瘤共生的根瘤菌属于Sinorhizobium meliloti和Sinorhizobium medicae两个种。张延明[12]采用此方法将309株大豆根瘤菌株分为10个基因种。MLST技术能够很好地鉴定根瘤菌属内不同种, 然而对同种内根瘤菌株进行划分的方法存在差异。Martinez[13]将一组可以根据生理或生化特性区别于同一物种其他菌株的细菌菌株划分为生物变体(biovars), 又根据植物宿主的共生能力和宿主范围的差异将细菌划分为共生变体(symbiovars)。关于生物变体和共生变体的划分在Rhizobium[14]、Bradyrhizobium[15]、Sinorhizobium[16, 17, 18]和Mesorhizobium[19]根瘤菌中均有报道。根瘤菌生物型是指同一种内的一组根瘤菌, 它们可以根据表型和在苜蓿品种上的特定共生模式而区别于其他菌株。生物型是在种内苜蓿品种水平上而不是种间寄主植物范围水平上, 根据表型生理生化特征和共生基因序列信息对根瘤菌进行的更为深入的鉴定, 是表型变体和共生变体的结合。属于同一物种的菌株表型是有差异的, 它们可能具有与共生相关的相同等位基因, 但会表现出不同的共生效应[20, 21], 这为同一种内基因差异的比较以及根瘤菌的分型奠定了基础。因此在分子生物学分型的基础上, 可以根据表型差异和在苜蓿品种上的共生模型将同种的根瘤菌株划分为生物型。近年来根瘤菌分型主要以表型生理生化特征或共生能力为依据, 将这两个因素结合对根瘤菌种内生物型的划分鲜见报道。本研究采用MLST技术对根瘤菌分型定种, 从表型和与苜蓿品种的共生效应两个角度对种内根瘤菌进行生物型划分, 为准确判断与苜蓿品种匹配的根瘤菌资源多样性以及为苜蓿与根瘤菌高效共生奠定基础。
采样时间为2014年5月和8月, 地点为甘肃省白银市会宁县会师镇牧草试验基地、武威市凉州区黄羊镇牧草试验站和甘肃农业大学兰州牧草试验站, 样地基本概况如表1所示。
| 表1 样地概况 Table 1 Geographical origin, ecological characteristics, and properties of sampled soils |
5个供试苜蓿品种均为紫花苜蓿, 已生长2年, 分别为国内选育品种甘农3号(M. sativa cv. Gannong No.3)和甘农9号(M. sativa cv. Gannong No.9)、地方品种陇中(M. sativa cv. Longzhong)和清水(M. sativa cv. Qingshui)、引进美国品种WL168HQ(M. sativa cv. WL168HQ), 见表1。
根瘤菌株培养采用YMA(yeast mannitol agar)培养基[22], 活化采用TY(tryptone yeast)培养基, 营养液采用Hoagland有氮和无氮营养液[23]。
于2014年5月(初花期)在每个苜蓿品种样地内随机取5株植株, 携带10 cm内根际土壤连根挖起, 抖落根际土壤并取样; 田间土壤取植株周围50 cm、深度20 cm内的土壤; 8月(成熟期)田间收集种子, 并清选。分别装于自封袋, 标记, 冰桶冷藏条件下带回实验室。清洗植株, 晾干表面明水后用无菌剪刀将植株分为花、茎、叶、根瘤、根表皮和根中柱; 将种子及以上组织各称取1 g置于无菌三角瓶内, 加碘伏(0.45%~0.55%)溶液震荡灭菌3 min[24], 无菌水冲洗5次, 加2 mL无菌水在研钵中研磨均匀[24]。分别称取10 g根际土壤和田间土壤置于无菌三角瓶内, 加入90 mL灭菌生理盐水, 充分震荡。将组织研磨匀浆和土壤悬浮液转入2 mL离心管离心(4000 r· min-1, 10 min), 用无菌水依次配置成10-3、10-4、10-5稀释液, 取0.2 mL稀释平板法涂抹至含刚果红的YMA培养基上, 28 ℃培养48 h, 进行内生菌株分离和纯化, 每个处理4个重复。纯化后的菌株经过形态特征和革兰氏染色检验, 于4 ℃保存于YMA培养基。
用上海生工细菌DNA提取试剂盒(SK8256)提取细菌基因组DNA, 方法参见试剂盒说明书。对细菌的16S rRNA以及atpD、glnII和recA持家基因片段进行PCR扩增。扩增引物参考Weisburg等[25]和Vinuesa等[26]的文献。所有引物均由上海生工生物公司合成, 扩增引物和PCR条件见表2。
| 表 2 16S rRNA, atpD, glnII和recA基因PCR扩增及测序引物 Table 2 Primers used for amplification and sequencing of 16S rRNA, atpD, glnII and recA genes |
供试菌株PCR扩增产物委托上海生工生物公司进行测序。通过EzBioCloud鉴定服务网站 (https://www.ezbiocloud.net/)[27]获得与目的序列同源性最高的模式菌株16S rRNA序列; 通过NCBI数据库 (www.ncbi.nlm.nih.gov/assembly)选择与目的序列同源性最高的模式菌株atpD、glnII和recA序列。用MEGA 6.0软件中的ClustalW进行序列相似性比对[28], 选用Neighbor-joining法(邻接法)进行UPGMA(非加权组平均法 unweighted pair-group method with arithmetic means)分析生成系统发育树, 发育树用Bootstrap法(自展法, 1000次重复)检验。
以YMA固体平板培养基为基础培养基, 对根瘤菌进行唯一碳氮源、抗逆性以及生理生化特性测定。唯一碳源测定项目包括苹果酸、肌醇、肌酸、甘露醇、蔗糖、葡萄糖、延胡索酸、琥珀酸、D-果糖和乳糖; 唯一氮源包括L-色氨酸、甘氨酸、精氨酸、L-组氨酸和苯丙氨酸; 染料抗性测定包括溴百里酚蓝、甲基红、甲基绿、溴酚蓝、亚甲基红、中性红、亚硝酸钠、孔雀石绿、溴甲基绿和刚果红; 唯一碳氮源利用和染料抗性测定设定浓度为1%; 抗生素敏感性测定包括红霉素、氯霉素、卡纳霉素、氨苄霉素、新霉素、链霉素和庆大霉素, 设置浓度梯度为5、50、100和300 μ g· mL-1; NaCl的浓度梯度为2%、4%和6%, 耐酸碱性测定pH为5、9和11, 生长温度包括8 ℃、37 ℃和40 ℃。生理生化试验包括淀粉水解实验、明胶水解实验、产硫化氢实验、VP实验、吲哚实验、接触酶反应、柠檬酸盐实验、3-酮基乳糖反应和BTB产酸产碱反应。菌株接种均设有3个阳性重复和1个阴性对照, 除生长温度测定实验在5~7 d后记录菌体生长状况外, 其余所有菌株接种后均于28 ℃恒温培养箱中培养, 24~48 h后记录菌体生长状况[29]。所有灭菌均采用121 ℃、26 min条件。
对根瘤菌的表型特征进行数值分类分析。将测定的表型性状结果按阳性记为“ 1” , 阴性记为“ 0” 进行编码后, 输入计算机。剔除全同性状, 利用NTSYS-PC 2.0 软件, 采用平均连锁法(UPGMA)生成聚类树状图。
种子处理、培苗以及根瘤菌菌液的制备方法参考李剑锋等[30]的文献。在幼苗生长第15天时, 在无菌条件下用移液枪将制备好的菌液加入幼苗根部, 每管4 mL, 加棉塞无菌培养。以不含根瘤菌的菌液为对照, 每个菌株4个试管作为重复。培养第22和30天, 在无菌条件下浇灌Hoagland无氮营养液, 30 d后去掉棉塞, 然后每隔5 d浇灌1次Hoagland无氮营养液[27], 每管4 mL。
接种效果测定:接种45 d后, 收获苜蓿植株并清洗干净, 用滤纸吸干水分。每管随机选取3株, 测算单株根瘤数、根瘤直径[28]、固氮酶活性[31]、根瘤等级[32]、单株有效根瘤重、株高、单株叶片数、地上鲜重、地上干重、叶绿素含量[33]、粗蛋白含量[32]、根长、地下鲜重和地下干重[34]。
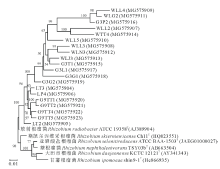
对所有细菌的16S rRNA基因进行部分序列测定, 结果表明, 78个纯化的菌株中, 仅20株菌属于根瘤菌属(图1)。其中内生菌株分别分离自根表皮(2)、根瘤(5)、根中柱(3)、茎(1)和种子(1)。8株非内生根瘤菌株来自根际土壤(3)和田间土壤(5)。如图1所示, 所有菌株均与模式菌株Rhizobium radiobacter ATCC 19358T聚在一起, 序列相似性为97.15%~98.72%。
对20个菌株的持家基因atpD、glnII和recA进行测序分析, 结果如图2所示。基于atpD(图2a)、glnII(图2b)和recA(图2c)系统发育树的拓扑结构表明菌株的系统发育地位不稳定, 虽然所有菌株在3个系统发育树中均形成8个分支, 但是菌株的进化地位存在差异。单个的持家基因系统发育分析不足以准确地对供试菌株进行分类。因此, 对于持家基因合并序列进行了MLST分析, 建立了20个菌株和参比菌株的系统发育树。 如图3和表3所示, 菌株G3T1、G3P2、WLG2、WLL3、WLJ3、WLL2、WLL4、LT2、WLL5、WTT4、WLN3和LP4与模式菌株R. radiobacter CCBAU 75229T聚在一起; 菌株G9TT1、G9TT5、G9TT2和LT3与模式菌株R. radiobacter CCBAU 75204T聚在一起; 菌株G9TT4、G3L1、G3G1和G3G2分别与模式菌株R. radiobacter CCBAU 75221T、R. radiobacter HAMBI 1814T、R. radiobacter CCBAU 73262T和R. radiobacter LMG 140T聚在一起。所有菌株序列相似性≥ 97%。经过持家基因合并序列MLST分析, 20个菌株被定种为R. radiobacter(表3)。
 | 图3 持家基因atpD、glnII和recA合并序列系统发育树Fig.3 Phylogenetic tree established by MLST from concatenated sequences of atpD, glnII, and recA |
| 表3 R. radiobacter菌株与模式菌株atpD、glnII和recA基因合并序列相似性 Table 3 Similarities of concatenated sequences of atpD, glnII and recA between the R. radiobacter isolates and the type strains |
所有根瘤菌在YMA培养基上呈圆形、光滑、乳白色透明状, 有黏质胞外多糖。菌体短杆状, 革兰氏染色反应阴性。对紫花苜蓿R. radiobacter菌株进行72项生理生化性状测定, 其中29项为全同性状, 对存在差异的43项性状以UPGMA聚类得到树状图谱, 如图4所示。在78%的相似性水平上, 所有菌株聚为7个群。群ⅰ 包括7个菌株(G3G1、WLJ3、WLL2、G3L1、G9TT4、G9TT5、G3T1), 群ⅳ 包括6个菌株(G9TT1、G9TT2、WLG2、WTT4、LT2、LT3), 群ⅱ (G3G2、LP4)和群ⅶ (G3P2、WLL5)各包含2个菌株。群ⅲ 、ⅴ 和ⅵ 分别包含菌株WLL4、WLN3和WLL3。43项差异性状的鉴别特征如表4所示。群ⅰ 、ⅱ 、ⅳ 和ⅶ 的菌株在唯一碳氮源利用、染料抗性、耐温性、耐盐性、抗生素耐性和生理生化反应方面表现均有不同, Shannon多样性指数(H)分别为5.41、2.43、4.08和2.77, 菌株表型多样性丰富。
 | 图4 20个R. radiobacter菌株表型数值分类聚类图Fig.4 Numerical taxonomy dendrogram based on the phenotypic features of 20 R. radiobacter isolates |
| 表4 数值分类特征表 Table 4 Characteristics distinguishing tested isolates |
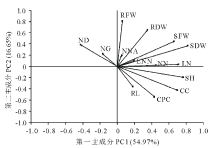
基于14个变量的主成分分析表明, 在标准化变量中第一主成分解释了总变异的54.97%, 第二主成分解释了16.65%的标准方差。如图5所示, 在第一主成分上, 地上干重(shoot dry weight, SDW)对苜蓿和根瘤菌共生效率的贡献率最大。因此选择地上干重进行多重比较, 分析菌株的共生效率差异。
不同根瘤菌株与苜蓿品种互作, 共生效果存在差异(表5)。菌株LP4接种甘农3号、菌株G9TT1、G9TT2、G9TT4和G9TT5接种甘农9号、菌株LT2接种陇中、菌株G3P2和G3T1接种清水苜蓿, 植株地上干重分别显著高于未接种处理CK(P< 0.05)。所有菌株接种WL168HQ苜蓿, 地上干重与CK差异不显著(P> 0.05)。为了比较根瘤菌株在各苜蓿品种上的共生效果, 当接种根瘤菌的苜蓿品种地上干重显著高于未接种处理CK时, 标记根瘤菌与苜蓿品种共生效应为A(P< 0.05), 与CK差异不显著时标记为B(P> 0.05), 显著低于CK时标记为C(P< 0.05)。以苜蓿品种为甘农3号、甘农9号、陇中、清水和WL168HQ的顺序, 将各根瘤菌株的共生效应结合, 得到根瘤菌株共生模式, 如表5所示。所有R. radiobacter菌株在5个苜蓿品种上存在6种共生模式, 分别为BBBBB(9)、BBBAB(4)、BABBB(4)、ABBBB(1)、BBABB(1)和BBAAB(1)。每个共生模式代表根瘤菌株与苜蓿品种之间的一种亲属关系。
| 表5 R. radiobacter菌株在5个苜蓿品种上的地上干重 Table 5 Shoot dry weight of R. radiobacter strains on five M. sativa varieties (mg· plant-1) |
具有相同表型, 并在苜蓿品种上表现相同共生效应的根瘤菌株代表一种生物型。本研究选择了5个苜蓿品种在同一物种内根据表型和共生模式进行根瘤菌生物型划分。结果表明20个R. radiobacter菌株被划分为14种生物型, 分离自各苜蓿品种的菌株具有丰富的多样性(表6)。来自甘农3号苜蓿的菌株被划分为生物型Ⅰ (G3G1和G3L1)、Ⅱ (G3G2)、Ⅲ (G3P2)和Ⅳ (G3T1)。来自甘农9号苜蓿的菌株被划分为生物型Ⅴ (G9TT1和G9TT2)和Ⅵ (G9TT4和G9TT5)。来自陇中苜蓿的菌株被划分为生物型Ⅶ (LP4)、Ⅷ (LT2)和Ⅸ (LT3)。来自WL168HQ苜蓿的菌株被划分为生物型Ⅰ (WLJ3和WLL2)、Ⅸ (WLG2)、Ⅹ (WLL3)、Ⅺ (WLL4)、Ⅻ (WLL5)、Ⅹ Ⅲ (WLN3)和Ⅹ Ⅳ (WTT4)。WL168HQ、甘农3号和陇中苜蓿内生物型多样性丰富, Shannon多样性指数H分别为2.03、1.39和1.10, 甘农9号和清水苜蓿多样性单一, H为0。
| 表6 20株R. radiobacter菌株基于表型和共生模式的生物分型 Table 6 Biotyping of 20 R. radiobacter isolates based on phenotype and symbiotic pattern |
NN(单株结瘤数), ENW(有效根瘤重), ND(根瘤直径), NG(根瘤等级), NNC(根瘤固氮酶活性), LN(单株叶片数), SH(株高), RL(根长), SFW(地上鲜重), RFW(地下鲜重), SDW(地上干重), RDW(地下干重), CC(叶绿素含量)和CPC(粗蛋白含量)。NN (nodule number), ENW (effective nodule weight), ND (nodule diameter), NG (nodule grade), NNC (nodule nitrogenase activity), LN (leaf number), SH (shoot height), RL (root length), SFW (shoot fresh weight), RFW (root fresh weight), SDW (shoot dry weight), RDW (root dry weight), CC (chlorophyll content) and CPC (crude protein content).
本研究从甘肃省3个栽培区域5个紫花苜蓿品种的不同部位分离得到78株细菌, 经16S rRNA基因部分序列测定有20株菌属于Rhizobium。持家基因MLST分析为种内根瘤菌株系统发育地位差异研究提供了依据, 在分子分型以及新种的鉴定方面应用广泛[38, 39]。已有研究应用此方法将紫花苜蓿根瘤菌鉴定为Sinorhizobium fredii、S. xinjiangense和S. medicae[29]以及R. leguminosarum[40], 而本研究持家基因合并序列MLST分析表明20株根瘤菌属于R. radiobacter。这进一步证实了16S rRNA序列分析结果, 也更详细地阐明了供试菌株的系统发育地位。与合并序列系统发育树状结构相比(6个分支), 在由atpD、glnII和recA基因建立的系统发育树上, 菌株分散在8个不同的进化分支中。与recA和合并序列MLST系统发育树不同, 在atpD系统发育树中, 菌株WTT4和WLL5与其余菌株系统发育关系较远, 与模式菌株R. radiobacter CCBAU 73262T形成独立的分支。同样的, 在glnII系统发育拓扑结构中, 菌株WLL3、WTT4、G3L1和WLL5与模式菌株R. radiobacter CCBAU 75204T聚在一起, 远离其余菌株形成独立分支。这种不确定的系统发育地位也许是受到水平基因转移现象的影响[16]。
研究表明基因型的改变会导致可遗传的变异, 然而由它所引起的表型改变是自然选择的底物[41]。相同基因型的菌株表现出不同的表型特征, 这可能是菌株在多个不同的环境中生存的结果[41]。本研究20个R. radiobacter菌株中有7种表型, 说明同一个种的根瘤菌表型多样性丰富[20]。表型的形成过程很复杂, 这个过程会影响新的和有益的表型的首次产生, 以及进化适应和创新的可能性[42]。表型的多样性因此也会导致根瘤菌株对苜蓿品种共生适应性的差异。
同一物种的根瘤菌株在5个苜蓿品种上共生效应表现存在差异。就地上干重而言, 所有菌株接种WL168HQ苜蓿均与未接种处理CK差异不显著(B)。菌株接种甘农3号、甘农9号、陇中和清水苜蓿地上干重显著高于CK(A)、与CK差异不显著(B)或显著低于CK(C), 共生效应表现多样。共生效应表现A的菌株在这4个苜蓿品种上表现出强的共生能力和适应性, 为根瘤菌与苜蓿品种间的亲属关系提供了证据。共生适应性越强, 亲属关系越近。6种共生模式代表菌株与苜蓿品种之间的6种亲属关系。共生模式BBBBB(9)表示菌株与5个苜蓿品种均无亲属关系; BBBAB(4)、BABBB(4)、ABBBB(1)和BBABB(1)表示菌株分别仅与清水、甘农9号、甘农3号和陇中苜蓿有亲属关系; 模式BBAAB(1)代表菌株与陇中和清水苜蓿均有亲属关系。根瘤菌与苜蓿品种之间的亲属关系与菌株的寄主来源没有关系。
同一种基因型根瘤菌中不同生物型的存在是菌株对不同豆科植物微环境适应性的体现[13]。同时, 由于共生模式可能与共生的质粒、岛屿或其他携带共生决定因素的染色体有关, 所以侧基因转移和共生信息基因丢失的假设也可以被用来解释同一根瘤菌基因型中生物型的出现[13, 16]。根瘤菌生物型由苜蓿品种和根瘤菌共同决定, 这也使得对种内根瘤菌划分成为可能。R. radiobacter基因型根瘤菌株在5个苜蓿品种上被划分为14个生物型, 每个生物型具有特定的表型特征和共生模式。不同苜蓿品种间根瘤菌生物型的种类有差异, WL168HQ、甘农3号和陇中苜蓿内根瘤菌生物型多样性很丰富。甘农9号和清水苜蓿多样性指数为0, 多样性单一, 这可能与数量太少有关。就生物型内苜蓿品种的多样性而言, 生物型Ⅰ (H=0.69)包含来自甘农3号和WL168HQ苜蓿的菌株, 生物型Ⅸ (H=0.35)包含来自陇中和WL168HQ苜蓿的菌株, 这两个生物型内苜蓿品种多样性丰富。其余12个生物型(H=0)多样性单一。根瘤菌生物型的划分对与苜蓿品种匹配的根瘤菌资源的准确判断具有指导意义。除了通过根瘤菌表型和与苜蓿品种的共生模式划分根瘤菌生物型之外, 应进一步利用根瘤菌和苜蓿品种基因组的研究结果加深对生物型的理解。
同物种根瘤菌株根据表型和共生效应可以划分为不同的生物型。WL168HQ、甘农3号和陇中苜蓿品种内生物型多样性丰富, 甘农9号和清水苜蓿生物型多样性单一。
The authors have declared that no competing interests exist.
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|
| [30] |
|
| [31] |
|
| [32] |
|
| [33] |
|
| [34] |
|
| [35] |
|
| [36] |
|
| [37] |
|
| [38] |
|
| [39] |
|
| [40] |
|
| [41] |
|
| [42] |
|